操作流程
分子优化操作流程如操作流程所示。
|
操作步骤 |
说明 |
|---|---|
|
需要完成相关订购后才可创建分子优化作业。 |
|
|
在AI科学计算平台创建新空间,并在空间范围内上传数据、创建作业等。 |
|
|
支持数据的添加、导入、引用、复制、删除等操作。 |
|
|
通过资产市场页面创建。 |
|
|
查看运行结果、收藏结果、分子详情、作业信息、聚类分析等。 |
步骤一:购买盘古药物分子大模型
- 进入AI科学计算平台后,左上角“单击”用户名,选择“资源订购”。
图1 资源订购
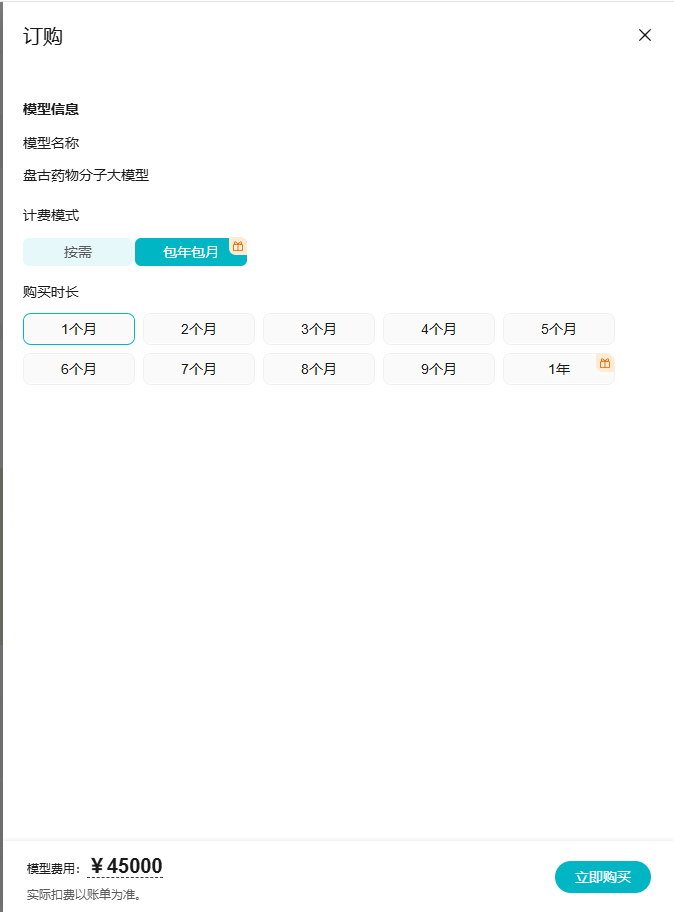
- 订购盘古药物分子大模型,可选择按需或者包周期。
图2 订购盘古药物分子大模型
- (可选)订购AI制药API套餐包。
- 可正常创建药物作业和提交属性模型作业。
图3 正常创建模型作业
步骤二:创建空间
您可以在AI科学计算平台创建新空间,并在空间范围内上传数据、创建作业等。“空间列表”展示了当前用户有权限访问的空间。
创建新空间时可直接创建一个OBS桶,也可使用已有的桶。
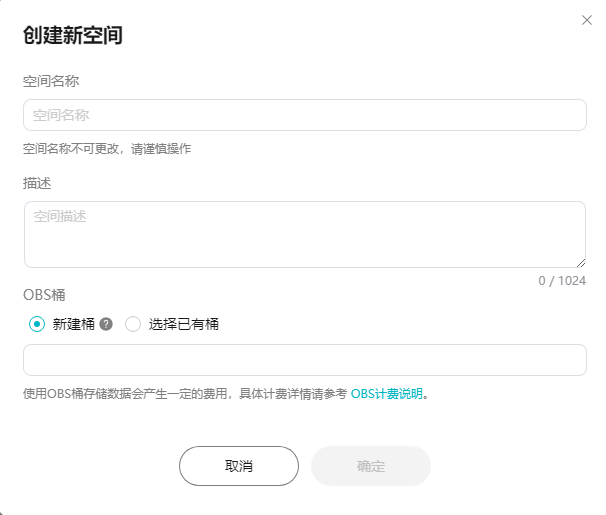
创建桶的命名遵循obs桶命名规则:
- 需全局唯一,不能与已有的任何桶名称重复,包括其他用户创建的桶。用户删除桶后,立即创建同名桶或并行文件系统会创建失败,需要等待30分钟才能创建。
- 长度范围为3到63个字符,支持小写字母、数字、中划线(-)、英文句号(.)。
- 禁止两个英文句号(.)相邻,禁止英文句号(.)和中划线(-)相邻,禁止以英文句号(.)和中划线(-)开头或结尾。
- 禁止使用IP地址。
步骤三:添加数据
在添加数据前,您可以创建存储数据的文件夹,将数据存储在对应的文件夹中。如果已有存储数据的文件夹,可以直接添加数据。
(可选)新建文件夹
- 单击空间名称,并选择“数据”。
- 单击“新建文件夹”,创建文件夹。
图6 新建文件夹
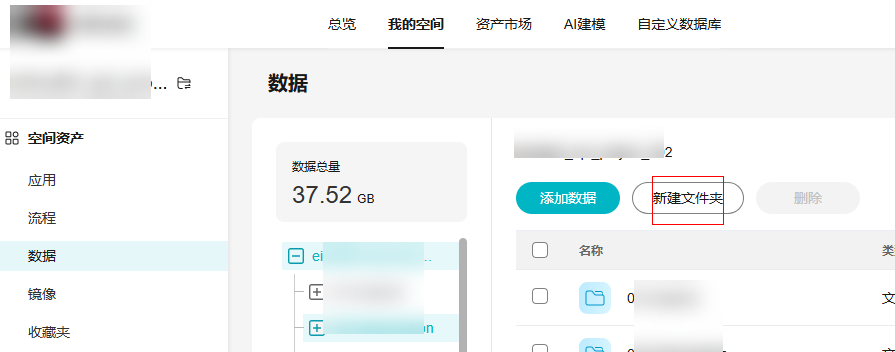

文件夹名称命名规则:
- 支持创建单个文件夹和多层级的文件夹。
- 单个斜杠(/)表示分隔并创建多层级的文件夹。
- 单个文件夹名称不能包含以下字符: \ / : ; * ? < " > |。
- 文件夹的绝对路径总长度不能超过1023字符。
- 文件夹名称不能以英文句号(.)、斜杠(/)或空格开头或结尾。
- 不能包含两个以上相邻的斜杠(/)。
- 文件夹名称不能以中划线(-)开头。
- 多层文件夹不能超过45层。
- 数据列表按照先文件夹后文件的顺序排列。文件夹默认为首字母排序,文件按照时间顺序排序,但始终在文件夹后面显示。
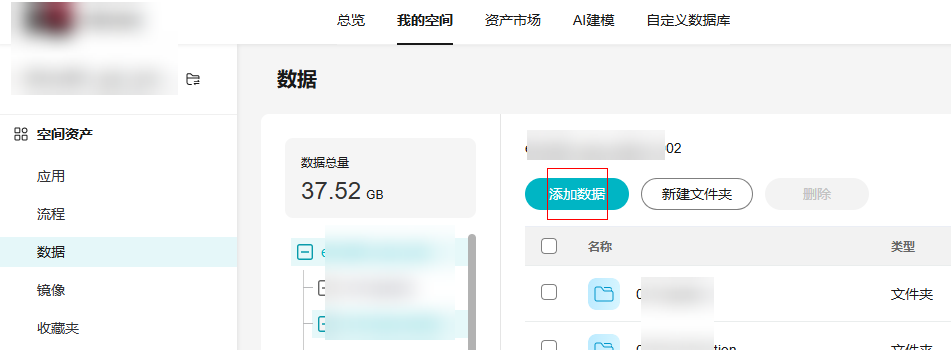
上传数据
- 在“数据”页签,选择数据需要上传的文件夹并单击“添加数据”,或者直接单击“添加数据”。
图7 添加数据
- 在添加数据页面,可以直接将文件拖拽至上传文件区域,也可以单击“拖拽或点击上传文件”区域,上传待分析数据。
图8 上传文件
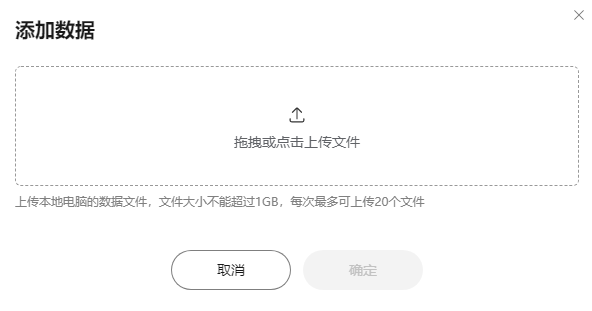
- 数据上传成功后,单击“确定”。

上传文件大小不能超过1GB。
更多添加数据方式,请查看《用户指南》手册“添加数据”章节。
步骤四:创建药物作业
分子优化基于盘古药物分子大模型,以参考化合物为起点,针对给定的期望理化性质,得到性质更优、结构新颖、与靶点蛋白亲和力高的化合物。
- 在“资产市场 > 小分子药物设计”页面,单击“分子优化”功能卡片的“立即使用”。
图9 立即使用

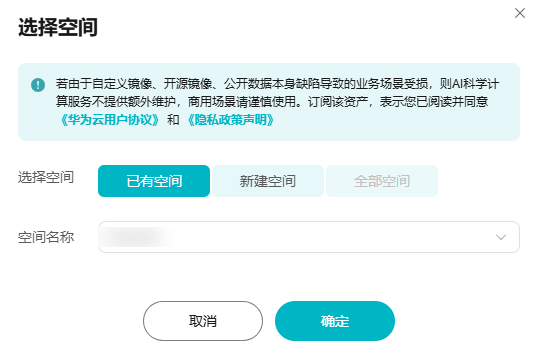
- 选择空间后,单击“确定”进入配置页面。支持在已有空间选择空间或者新建空间。
图10 选择空间
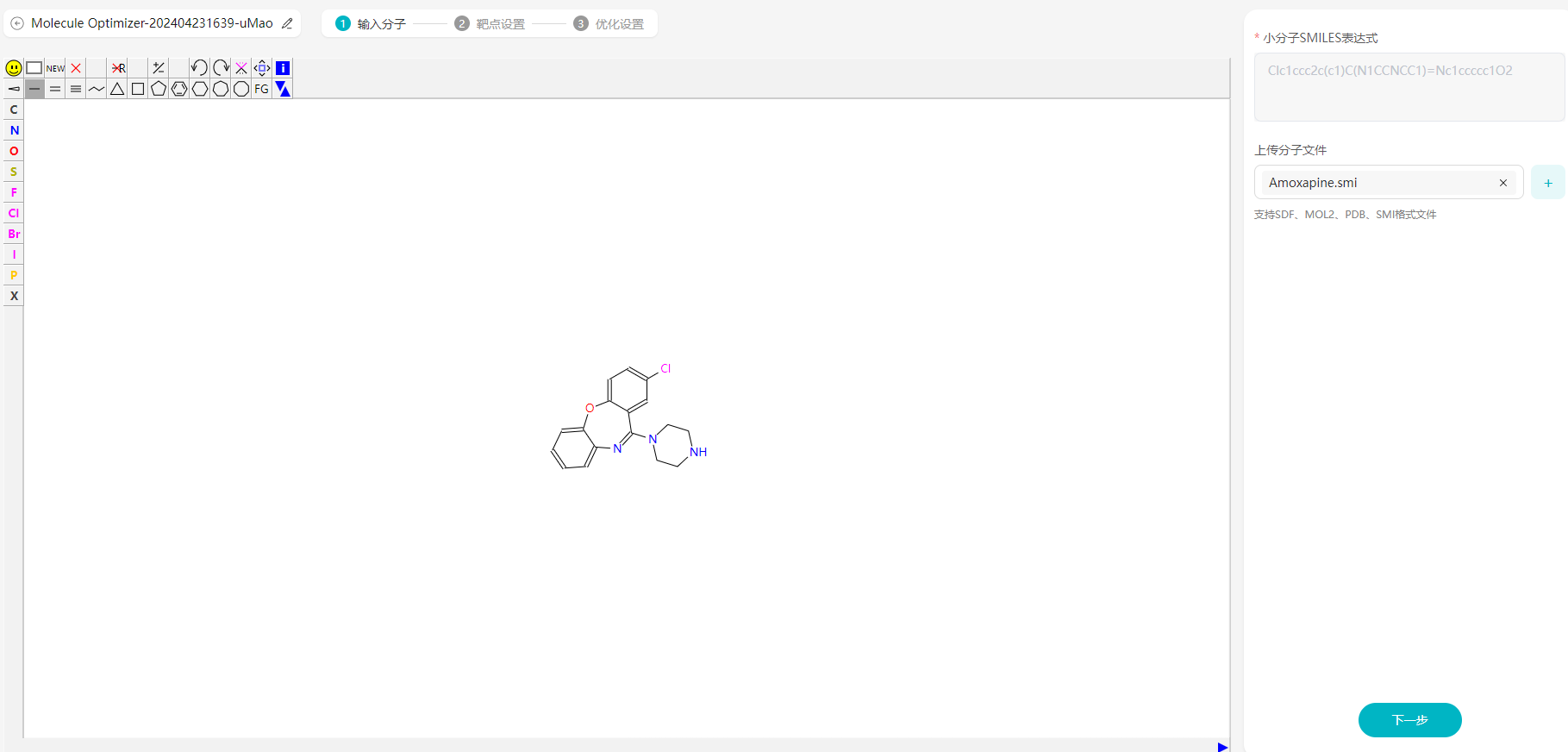
- 在白框内输入需要优化的小分子SMILES表达式或者上传分子文件。
图11 输入分子
- 单击“下一步”,进入靶点设置(可选步骤)。
如果需要设置靶点,并且将对接结合能作为一个约束条件进行优化,需要进行配置。最多可添加2个靶点。在添加靶点后,会进行单分子预对接,可查看Top1的对接打分与构象。
如果不需要设置靶点,此步骤可以进行省略,如果设置了靶点,作业运行时间会加长。
图12 靶点设置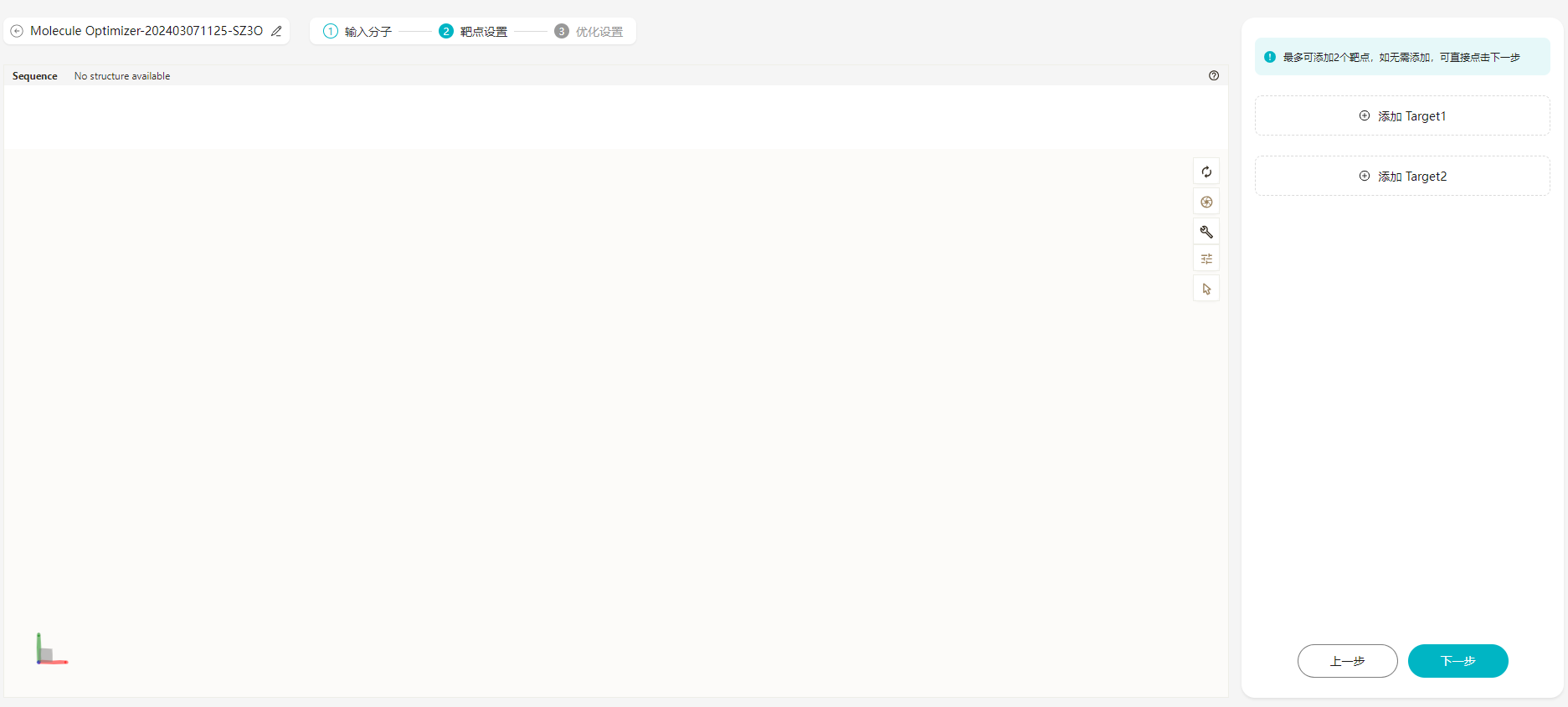 图13 Target设置
图13 Target设置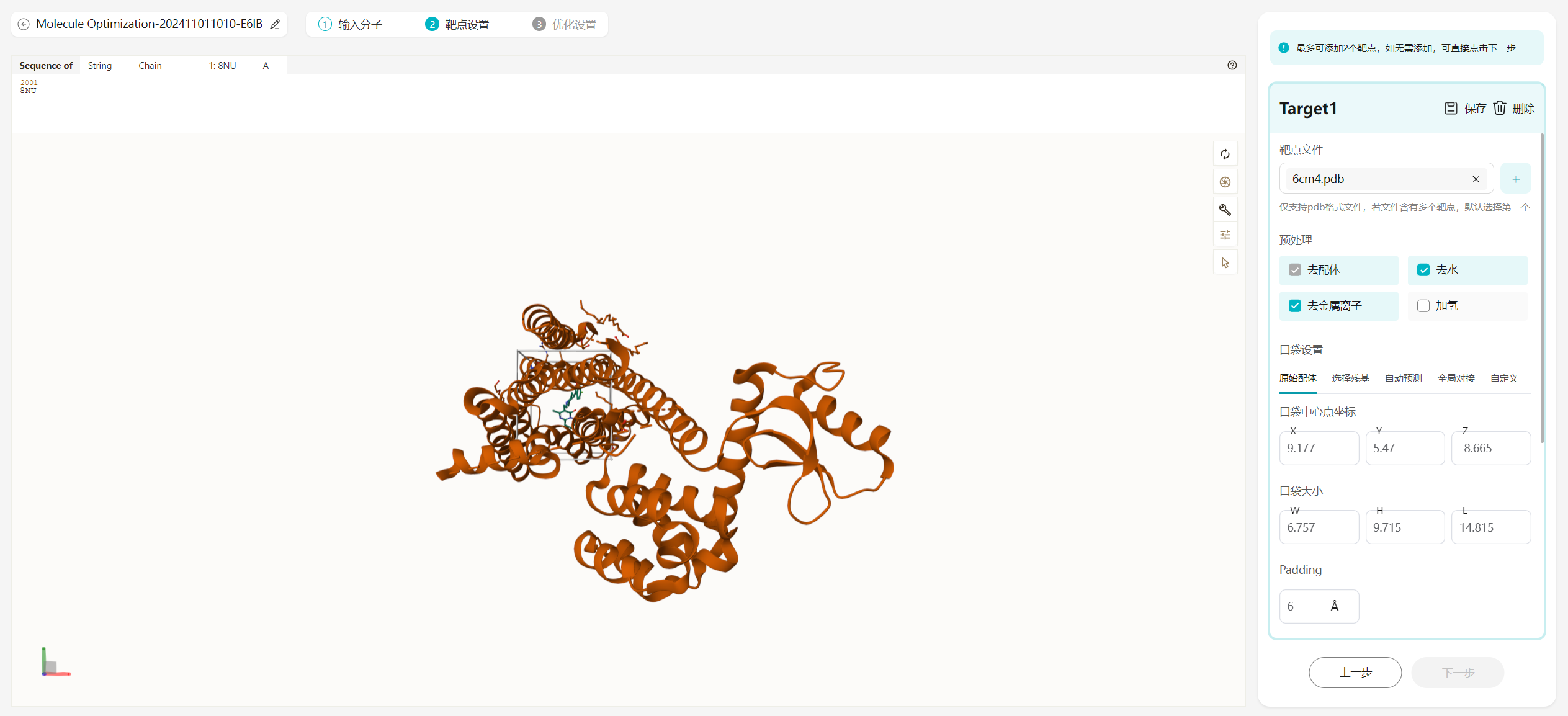 图14 靶点设置完成
图14 靶点设置完成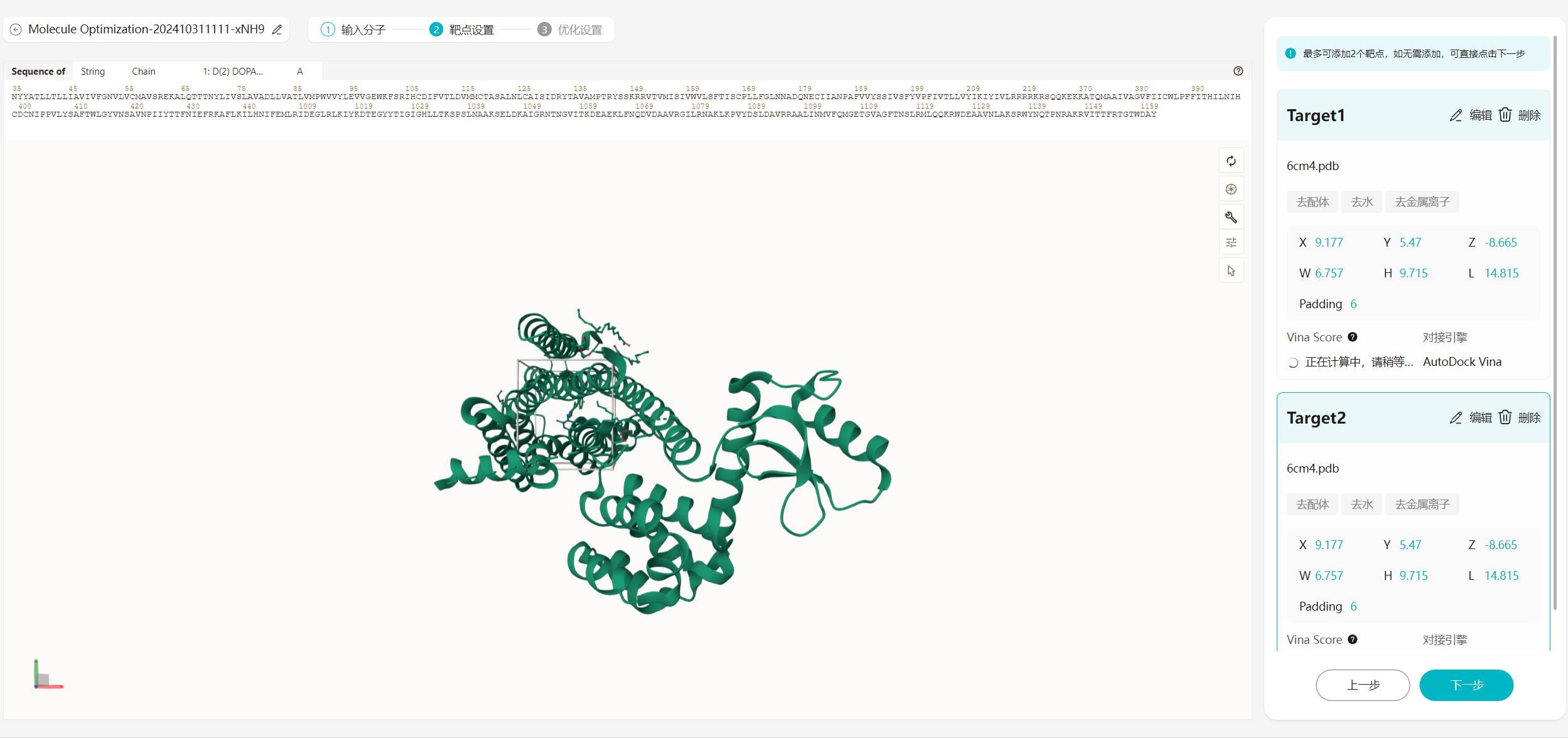
- 单击“下一步”,进入优化设置页面。
图15 优化设置页面(1)
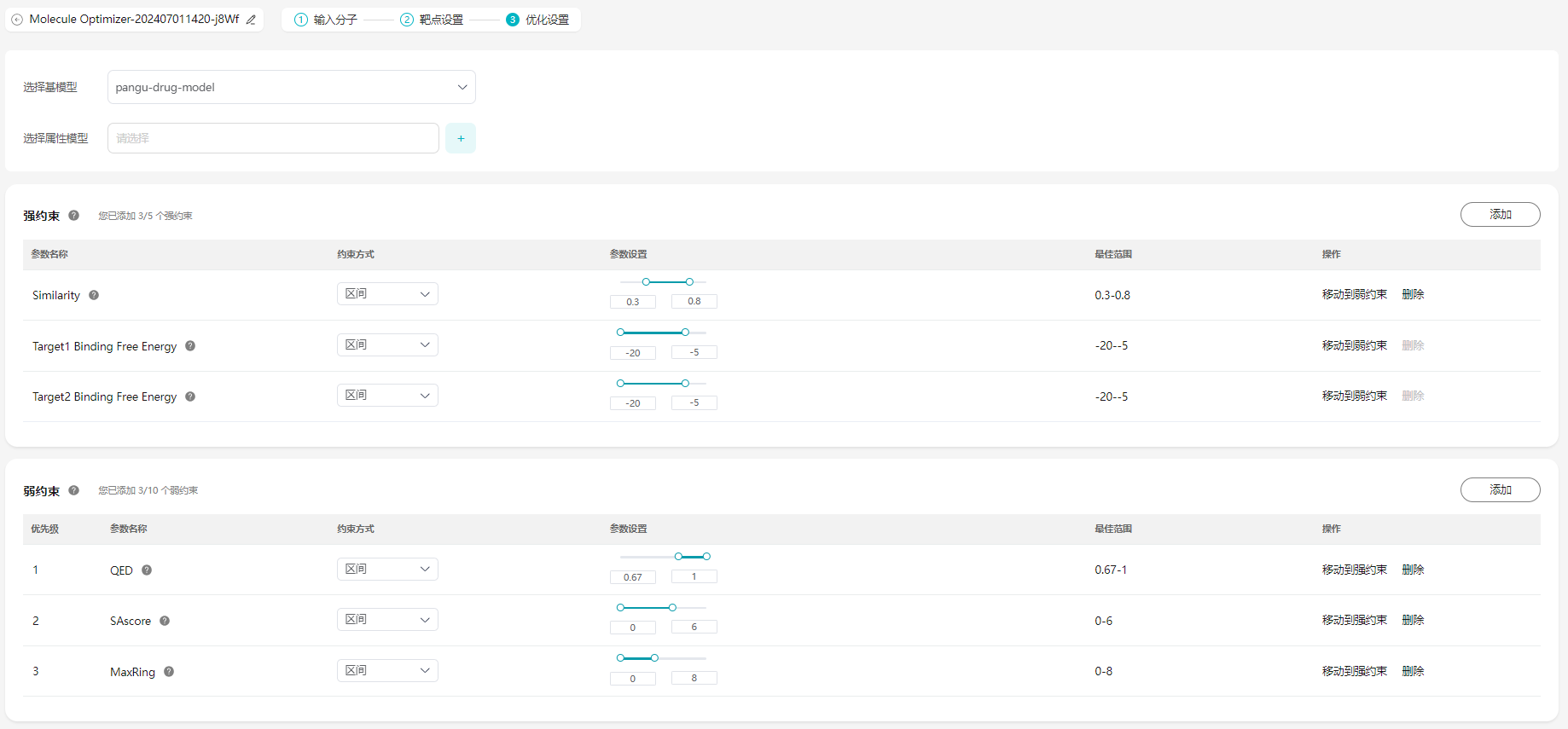 图16 优化设置页面(2)
图16 优化设置页面(2)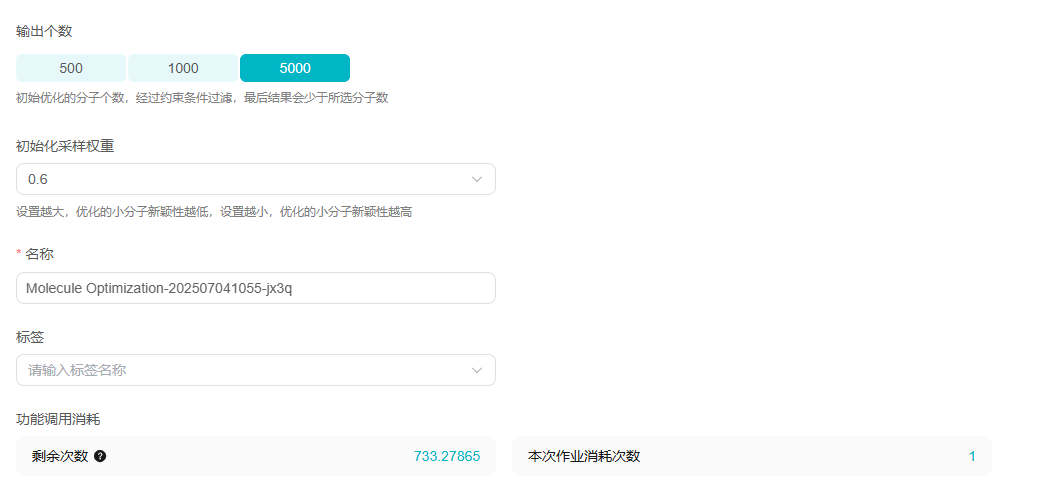
- 选择基模型:支持选择基模型。如果选择的基模型是非官方盘古药物大模型,则约束条件不支持官方机器学习属性,只支持以该基模型创建的属性模型作为约束条件。基模型列表见AI建模。
- 选择属性模型:选择AI模型。如果需要创建模型,可参考属性模型。一次最多可以选10个模型属性。属性模型的基模型必须与上一步所选择的基模型一致。
- 优化强约束、弱约束的参数和参数值。
- 强约束:优化后的小分子必须要满足的约束条件。强约束用于严格筛选输出分子,若分子不满足其中任何一条约束则会被直接丢弃。强约束条件个数1~3个最佳,不宜过多,过多的强约束会导致输出结果数量少于预期或者没有分子生成。如果设置的官能团结构太大,建议放到弱约束,因为这样设置会使模型可探索的区间比较小,导致可能没有结果生成。如果分子较难优化,优化后的分子数过少,建议可以适当放宽强约束的条件设置,比如相似度可以放宽到0.3~1.0。如果分子较易优化,优化后的分子相似度较高,新颖性较低,建议可以适当收紧强约束的条件设置,比如相似度可以收紧到0.3~0.7等等,正常情况下相似度设置按照默认即可。
- 弱约束:优化后的小分子不必要满足的约束条件。弱约束用于打分排序最终结果,输出分子会按照弱约束顺序尽可能多地满足所有弱约束;不满足弱约束的结果依然会被保留在结果中,对于一个比较关注且重要的分子属性,建议放到强约束条件设置里,因为弱约束不会做过滤。
如果进行了“靶点设置”,会自动加上相应靶点的“Binding Free Energy”才会计算对接结合能,并将对接结合能作为约束条件进行分子优化。
您可以通过“添加”来添加新的约束条件,也可以在操作列单击删除图标,删除约束参数。每个约束参数的含义参考小分子药物设计相关参数。
强约束最多选择5个,仅Substructure和Interaction参数可重复选择,其余不可以;弱约束最多选择10个,可以重复选择,弱约束的初始权重与弱约束的顺序有关,弱约束条件越靠前则初始权重越大。
- 输出个数:选择输出个数,目前支持500、1000、5000。输出个数越多,任务时间越长。
- 名称:可修改,修改后左上角也同步修改。长度为5~64个字符;仅可以使用字母、数字、下划线“_”、中划线“-”和空格;首位只能以数字或字母开头。
- 初始化采样权重:设置越大,优化的小分子新颖性越低,设置越小,优化的小分子新颖性越高。
- 选择属性模型:选择AI模型。如果需要创建模型,可参考AI建模。一次最多可以选10个模型属性。
- 标签:设置任务标签。
- 功能调用消耗:运行一次作业会消耗对应的调用次数。

优化后的小分子在满足强约束条件的基础上,会根据满足弱约束条件的权重总和以及与参考小分子的相似度来打分并进行排序。在初始化权重的基础上,每个约束所占的权重,会在每一轮的分子优化迭代中,根据所满足的约束来进行动态调整。比如说约束条件1,在分子优化迭代中比较容易满足,那么该条件的权重会降低,如果不容易满足,该条件的权重会升高。
如果需要设置官能团的约束,可以在约束条件中设置“Substructure”,然后选择“包含”或者“排除”,包含官能团或者去除官能团条件,设置1个官能团数为佳。在官能团设置中,可以单击蓝色按钮来设置生长方向,即所优化的分子只会往所标记的方向生长。如果在一个Substructure约束条件里面添加多个官能团,则官能团之间的关系是“或”,即生成的分子满足其中一个官能团即可。如果添加多个Substructure约束,则官能团之间的关系是“和”,即生成的分子都会满足这几个官能团。
如果需要设置相互作用力的约束,可以在约束条件中设置“Interaction”,然后选择“包含”或者“排除”,包含相互作用力或者去除相互作用力。在相互作用力约束条件设置中,可以选择相应的靶点、氨基酸和相互作用力,相互作用力支持H bond,Hydrophobic,Salt Bridge,Pi Stacking,Pi Cation。如果在一个Interaction约束条件里面添加多个相互作用力,则相互作用力之间的关系是“或”,即生成的分子满足其中一个相互作用力即可。如果添加多个Interaction约束,则相互作用力之间的关系是“和”,即生成的分子都会满足这几个相互作用力。
约束方式,包含“区间”,“最大化”和“最小化”种方式,“区间”指的是所选择的属性包含在所设置的参数设置区间内。“最大化”指的是所设置的属性越大越好,而不只是限定在某区间内,当无法判断属性应该设置在什么区间,但是属性越高,成药性越好,可以设置属性设置条件为最大化,这个只能在弱约束里面进行设置。“最小化”指的是所设置的属性越小越好,与最大化相反,也是只能在弱约束里面进行设置。
相似度分数,是利用ECFP4分子指纹计算优化后分子与原始分子的Tanimoto相似性。设置了禁止优化列表,禁止以高毒性为优化目标的属性优化,列表为:hERG Blockers,H-HT,DILI,AMES,Skin Sensitization,Carcinogencity,Eye Irritation,Eye Corrosion。
- 单击“提交”。
步骤五:获取结果
- 查看运行结果。
- 可以以列表的形式查看分子优化的作业,单击左上角“下载”,下载分子优化的结果或者分子3D构象。如果分子设置了靶点,可以下载小分子或复合物,若分子未设置靶点,只能下载小分子。小分子支持SDF和PDB格式,复合物只支持PDB格式。
- 单击
 可以收藏结果,收藏后可直接在收藏夹页查看。
可以收藏结果,收藏后可直接在收藏夹页查看。 - 分子优化对应的下游分析为分子优化和分子搜索,如果分子设置了靶点,可以选择自由能微扰进行下游分析,通过单击“下游分析”可以进行创建。
- 如果分子设置了靶点,可以单击“下载3D”下载优化后的小分子或者复合物,如果分子未设置靶点,单击“下载3D”只能下载小分子。小分子支持SDF和PDB格式,复合物只支持PDB格式。
- 如果添加了靶点,支持按照相互作用力进行高级筛选,单击
 进行条件配置。
进行条件配置。
图17 查看结果(1) 图18 高级筛选
图18 高级筛选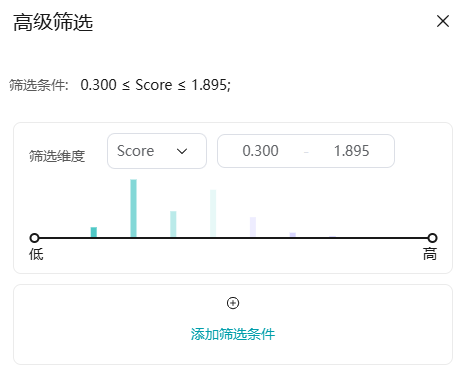
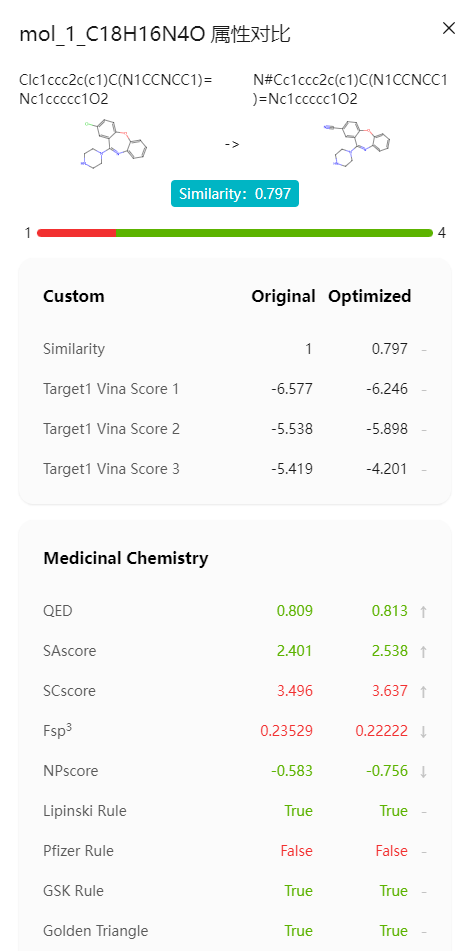
- 分子优化的结果可以查看原始配体和生成后的配体的结构以及属性值对比,可单击“对比”进行查看。可以直观地看到哪些属性变好了,哪些属性变坏了。
图19 属性对比
- 分子优化结果支持以卡片视图的形式进行查看,参考图21,在卡片视图中:
- 单击右上方的选择下拉框,可以选择分子的排序方式,将分子按照所选的排序方式进行展示。
图20 排序方式
- 单击每个分子卡片右上方的
 可以收藏结果,收藏后可直接在收藏夹页查看。
可以收藏结果,收藏后可直接在收藏夹页查看。 - 单击每个分子卡片右上方的
 ,可以选择“查看详情”、“查看3D”、“下游分析”、“下载3D”。
,可以选择“查看详情”、“查看3D”、“下游分析”、“下载3D”。
- 查看详情:单击查看详情,跳转至分子详情页。
- 查看3D:查看分子的3D视图。
- 下游分析:分子优化对应的下游分析为分子优化和分子搜索,如果分子设置了靶点,可以选择自由能微扰进行下游分析,单击“确定”即可创建。
- 下载3D:如果分子设置了靶点,可以单击“下载3D”下载优化后的小分子或者复合物,如果分子未设置靶点,单击“下载3D”只能下载小分子。小分子下载支持SDF和PDB格式,复合物下载只支持PDB格式。
- 每个分子卡片上会展示相应分子序号与对应的参数Vina Score(有靶点)、Similarity、Score、QED、SaScore
- Vina Score:代表分子如果添加了靶点,将会计算对接结合能,并按照Vina Score进行排序。
- Similarity:代表优化后的分子与原始分子的相似度。
- Score:代表优化后小分子的综合打分。
- QED:代表分子的成药性。
- SaScore:代表合成可及性分数,旨在评估分子的合成难易程度。

无靶点时显示:Similarity、Score、QED、SaScore。
单靶点时显示:Similarity、Score、QED、Vina Score。
双靶点时显示:Similarity、Score、Vina Score1、VinaScore2。
单击“查看3D”,可以看到分子的3D构象,如果设置了靶点,还可以看到优化后的小分子与靶点的结合构象。
如果上传了双靶点,可以通过切换
 来切换靶点,查看相应靶点和优化后分子的结合构象。如果设置了两个靶点会默认下载两个靶点的结果。
来切换靶点,查看相应靶点和优化后分子的结合构象。如果设置了两个靶点会默认下载两个靶点的结果。单击配体列表的
 可以收藏结果,收藏后可直接在收藏夹页查看。图22 查看3D图
可以收藏结果,收藏后可直接在收藏夹页查看。图22 查看3D图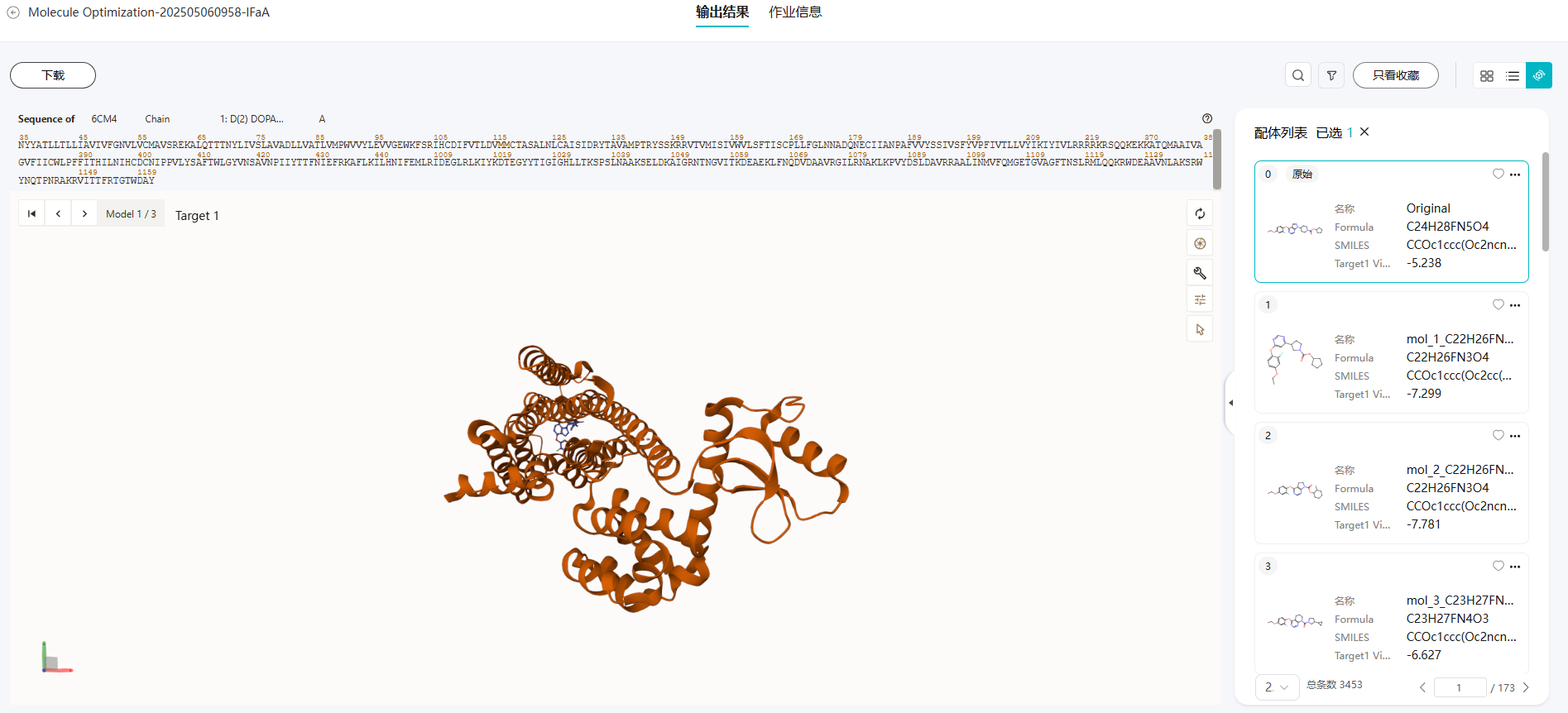
在查看3D的页面中,单击右侧的配体列表中,每个配体卡片右上角的
 ,可以查看:
,可以查看:- 查看详情:可以查看每个分子的属性信息,score和similarity。
- 查看属性:查看每个配体的分子属性。
- 查看2D相互作用图:查看配体中分子之间的2D相互作用图,并且可以进行图片下载。
- 下游分析:分子优化对应的下游分析为分子搜索和分子优化,如果分子设置了靶点,可以选择自由能微扰进行下游分析,单击“确定”即可创建。
- 下载3D:选择下载小分子或者复合物后,单击“确定”,小分子支持SDF和PDB格式,复合物只支持PDB格式。
- 单击右上方的选择下拉框,可以选择分子的排序方式,将分子按照所选的排序方式进行展示。
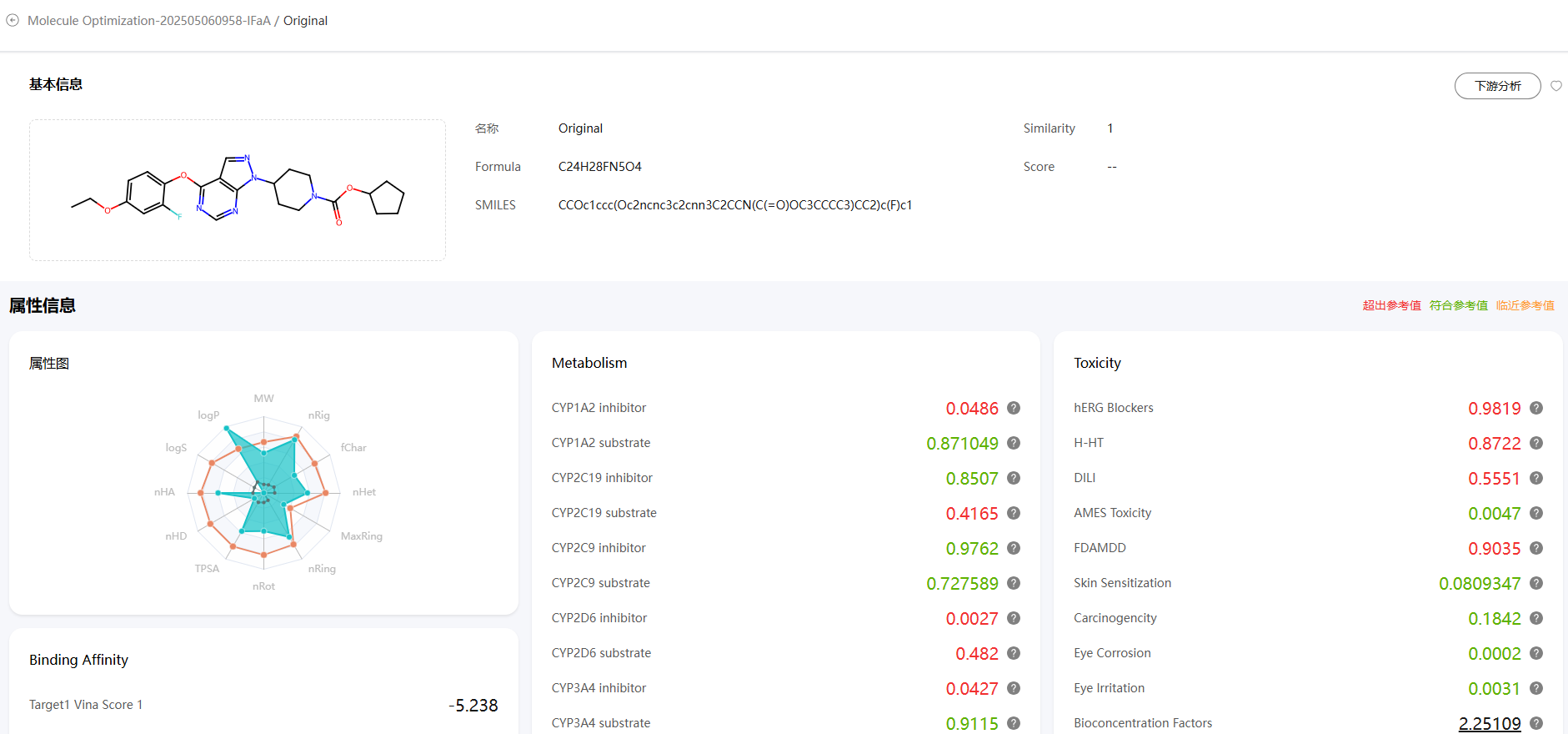
- 查看分子详情。
图23 查看分子详情
- 查看作业信息。
单击“作业信息”切换到作业信息页签,可以查看作业的分子信息、靶点信息与优化信息等。
图24 作业信息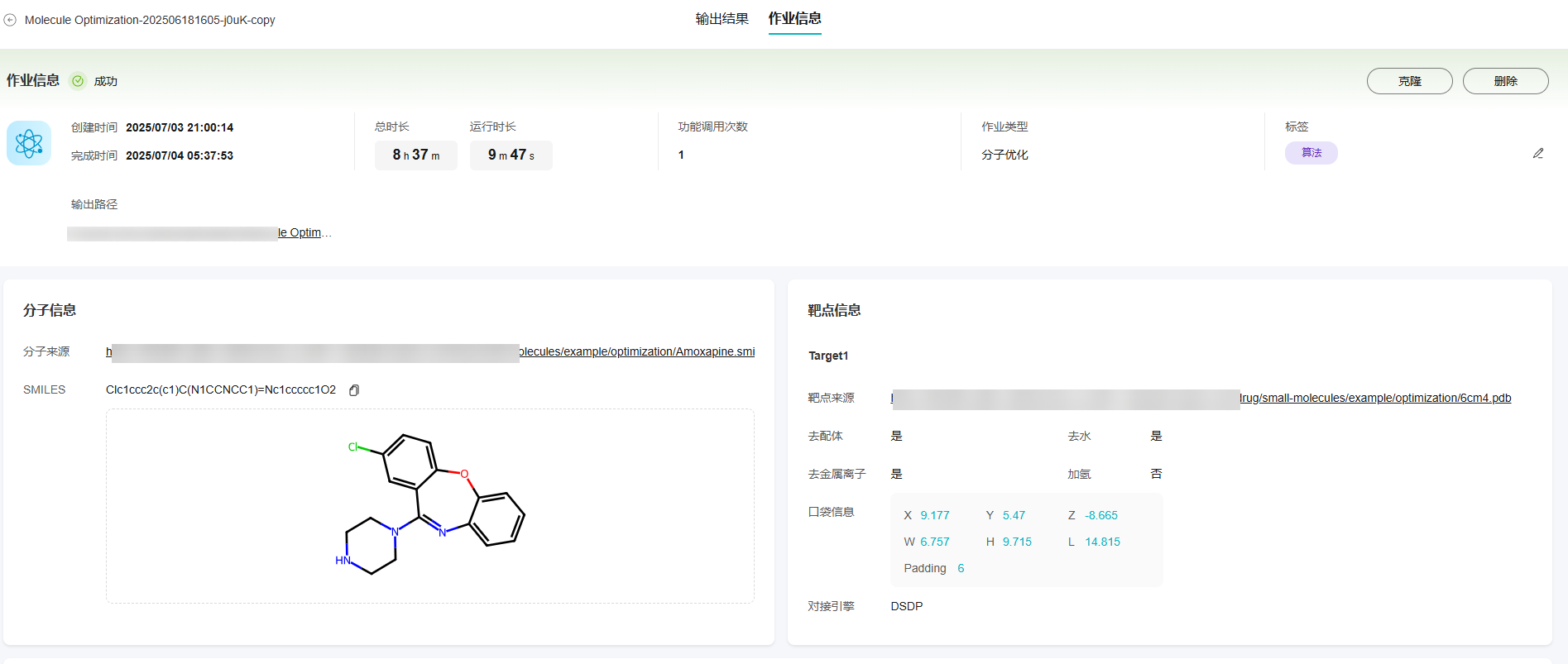
- 聚类分析
目前分子优化返回的结果小分子数较多,无法进行批量分析,通过一些聚类的辅助方式能更好地选择分子。从每个类里挑选出一两个分子进行后续分析和验证,提高分析的效率和分析质量。也可以通过聚类找出一些关键的骨架,来进行下游分析或者优化等。
- 在输出结果页面左上角单击“聚类分析”后,系统开始进行分析,同时显示“聚类分析中”。
图25 聚类分析
- 待聚类分析完成后,单击“查看聚类结果”。进入聚类结果页。
- 在聚类结果页面,可以查看每个聚类的分子数量等信息。
图26 查看聚类结果
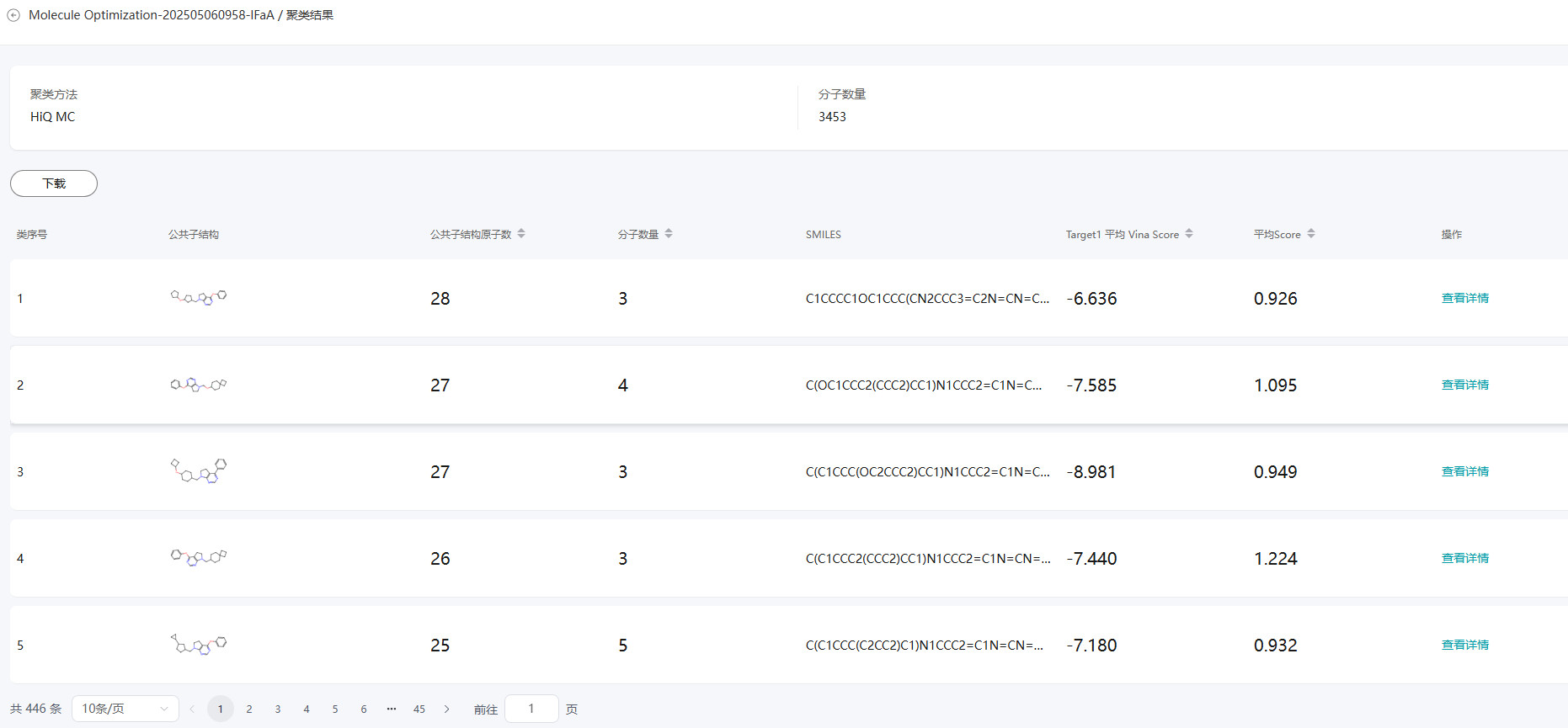
- 单击某个聚类的操作列的“查看详情”,即可进入聚类详情页面,聚类详情页支持以卡片、列表以及3D的形式查看。默认展示卡片页面,用户可自行进行切换。

- 每个结果页面只用进行一次聚类分析操作。
- 聚类结果是存成文件,如果文件被删或者获取不到的话会有警告, 聚类结果不存在。此时可以单击“重新聚类分析”。
- 如果聚类失败,根据提示失败原因解决问题后,可单击“重新聚类分析”。
- 在输出结果页面左上角单击“聚类分析”后,系统开始进行分析,同时显示“聚类分析中”。







