分子对接
分子对接基于华为云大算力,可根据靶点蛋白和小分子药物的3D结构,计算对接结合能,实现百万级别虚拟筛选。
- 在“资产市场 > 小分子药物设计”页面,单击“分子对接”功能卡片的“立即使用”。
图1 立即使用
- 选择空间后,单击“确定”进入配置页面。支持在已有空间选择空间或者新建空间。
图2 选择空间
- 在分子对接受体预处理页面,单击
 上传受体文件,进行受体蛋白预处理配置。
上传受体文件,进行受体蛋白预处理配置。
受体文件仅支持PDB格式,若文件中存在多个受体,默认只处理第一个。受体文件支持上传1个或者多个文件,最多支持输入20个受体文件,文件来源包含数据中心、PDB库、示例数据。
图3 上传受体文件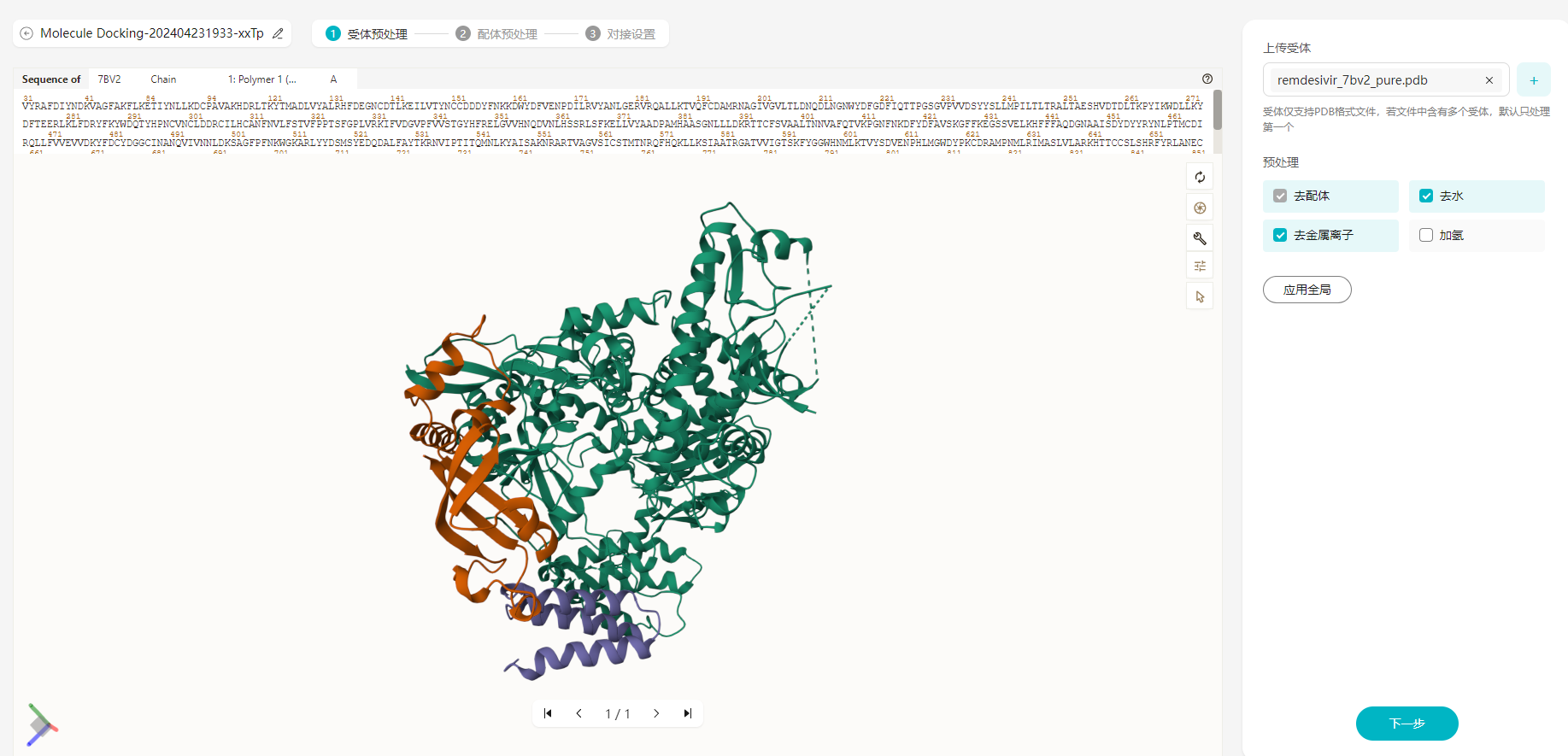
- 受体预处理参数配置。
- 去水:去掉受体蛋白里面的水分子。
- 去金属离子:去掉受体蛋白里面的金属离子。
去水、去金属离子参数默认是勾选状态,若上传的受体中包含配体,提交任务时系统会自动删除配体。
- 单击“应用全局”,可以对受体进行批量处理。
- 单击“下一步”,进入配体预处理页面。
- 单击“上传配体”后的
 ,选择配体文件。
,选择配体文件。
配体文件仅支持输入1个文件。
配体支持SDF、MOL2、PDB、 SMI格式文件,SMILES最多512字符,不能有中文或者空格,重原子不能超过200,配体分子需合法。
配体列表可显示总条数,但最多展示1000个小分子,对接时使用全量小分子。配体来源包含:数据中心、示例数据、公共库。且公共库预置了分子数据库,包含了陶术数据库、DrugSpaceX数据库、DrugBank数据库可以进行使用,单击“公共库”,可以进行选择。
图4 选择配体文件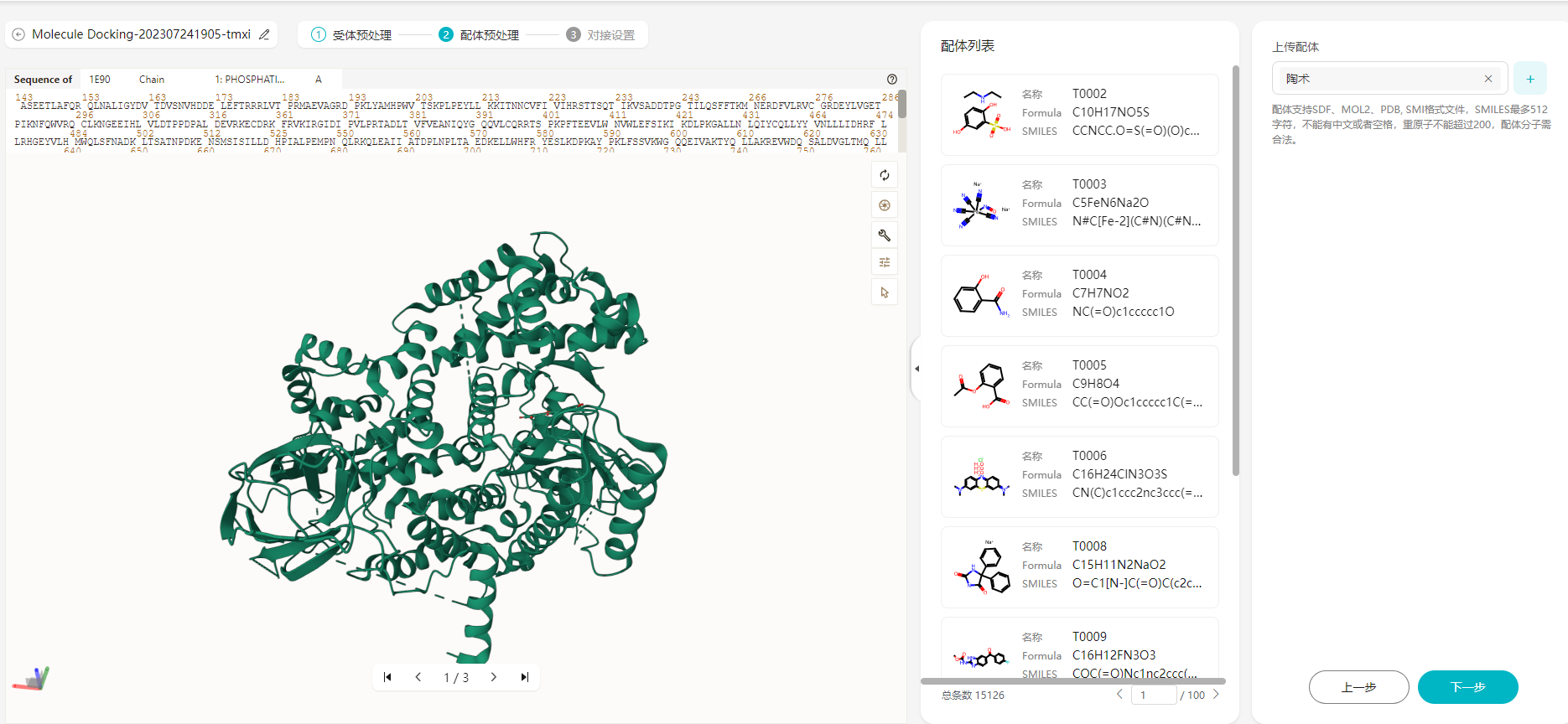
- 单击“下一步”,在对接设置页面配置相关参数。
图5 设置参数信息
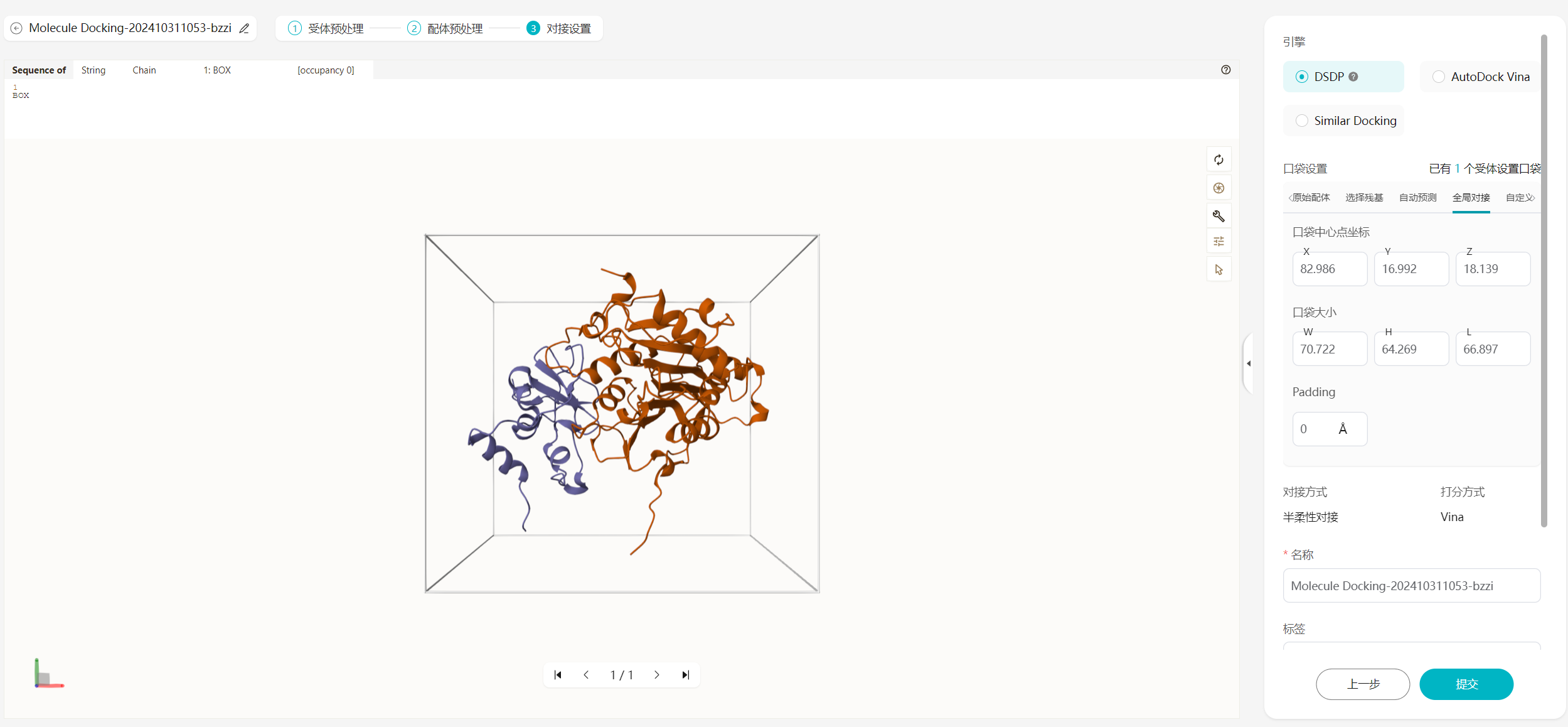
- 引擎:对接引擎支持DSDP,AutoDock Vina和Similar Docking。DSDP是由北大高毅勤团队研发的基于GPU加速的对接方法,支持全局对接。
- 配置受体口袋位置,如果包含多个受体文件,需要每个受体都进行口袋设置,才能单击提交。
- 功能调用消耗:每20000对会消耗一次功能调用。
- 配置成功后,单击“提交”。
- 查看分子对接结果。
如果是多受体对多配体,打开作业结果页面可以看到结合能二维矩阵,支持分别按照靶点和小分子进行排序。
图6 查看输出结果(1)
单击受体结合能框,跳转到单受体对多配体的结果表页面,可以下载全量及单条对接结果。
如果需要下载多个结果,可以选择结果后,单击左上角的“下载”。如果需要下载单条数据,单击数据操作列的“下载3D”即可。
图7 查看输出结果(2)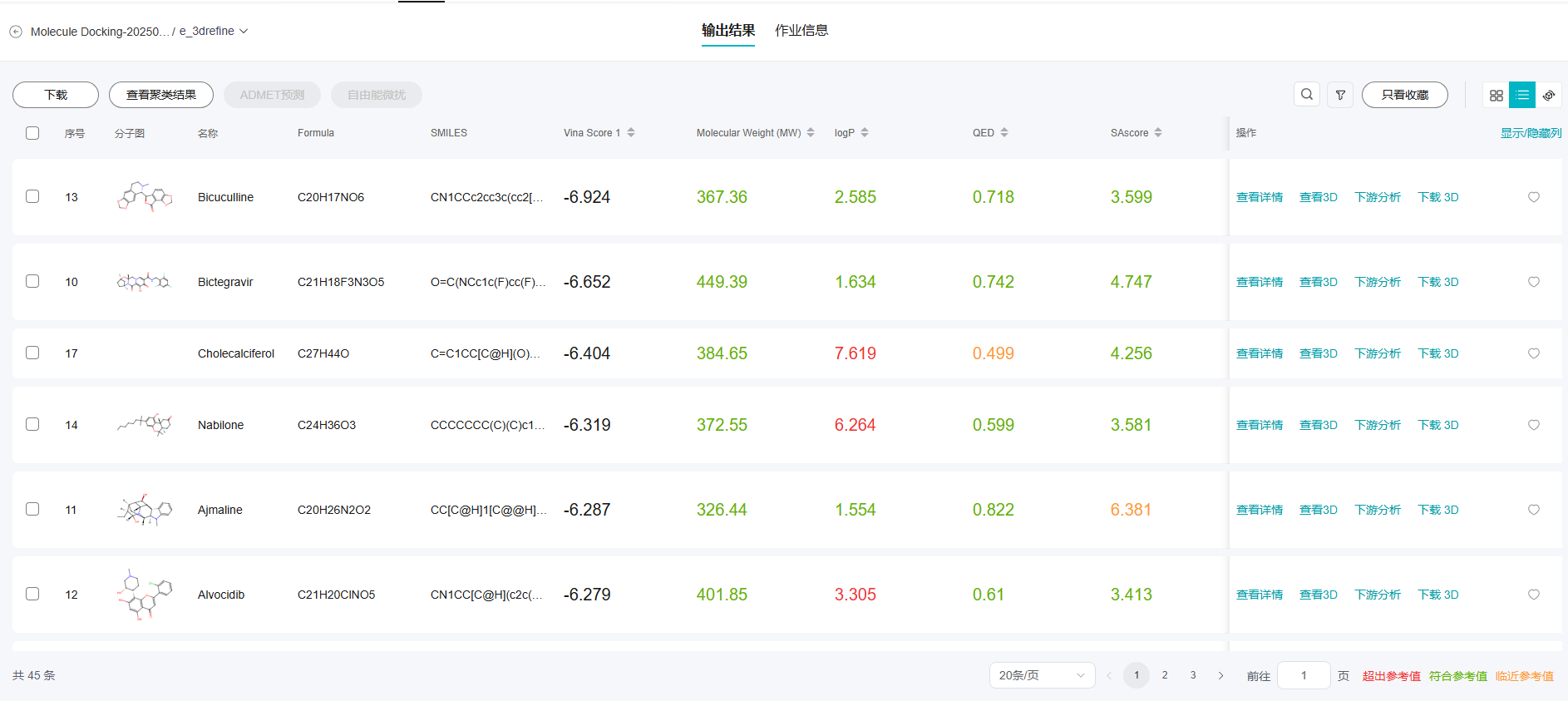
单击
 可以按照属性以及相互作用力进行高级筛选。图8 高级筛选
可以按照属性以及相互作用力进行高级筛选。图8 高级筛选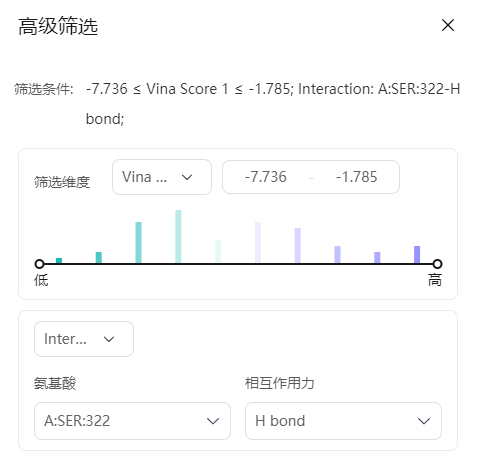
单击“查看3D”可以看到小分子和靶点的对接构象。
图9 查看3D 单击配体对比,可以计算对接后的构象与输入小分子的RMSD值和输入小分子和靶点的相互作用2D图。图10 设置配体对
单击配体对比,可以计算对接后的构象与输入小分子的RMSD值和输入小分子和靶点的相互作用2D图。图10 设置配体对 图11 配体对接结果图
图11 配体对接结果图 单击“查看2D相互作用图”,可以看到对接后的小分子构象与蛋白的2D相互作用图,并且支持相互作用2D图下载。图12 查看2D相互作用图
单击“查看2D相互作用图”,可以看到对接后的小分子构象与蛋白的2D相互作用图,并且支持相互作用2D图下载。图12 查看2D相互作用图
单击“下游分析”选择分子搜索、分子属性预测、分子优化或自由能微扰后,单击“确定”即可创建下游分析。
单击“下载3D”可以下载小分子或复合物,小分子支持SDF和PDB格式,复合物只支持PDB格式。
单击“查看详情”可以直观查看小分子的结合能和属性信息。
图13 查看详情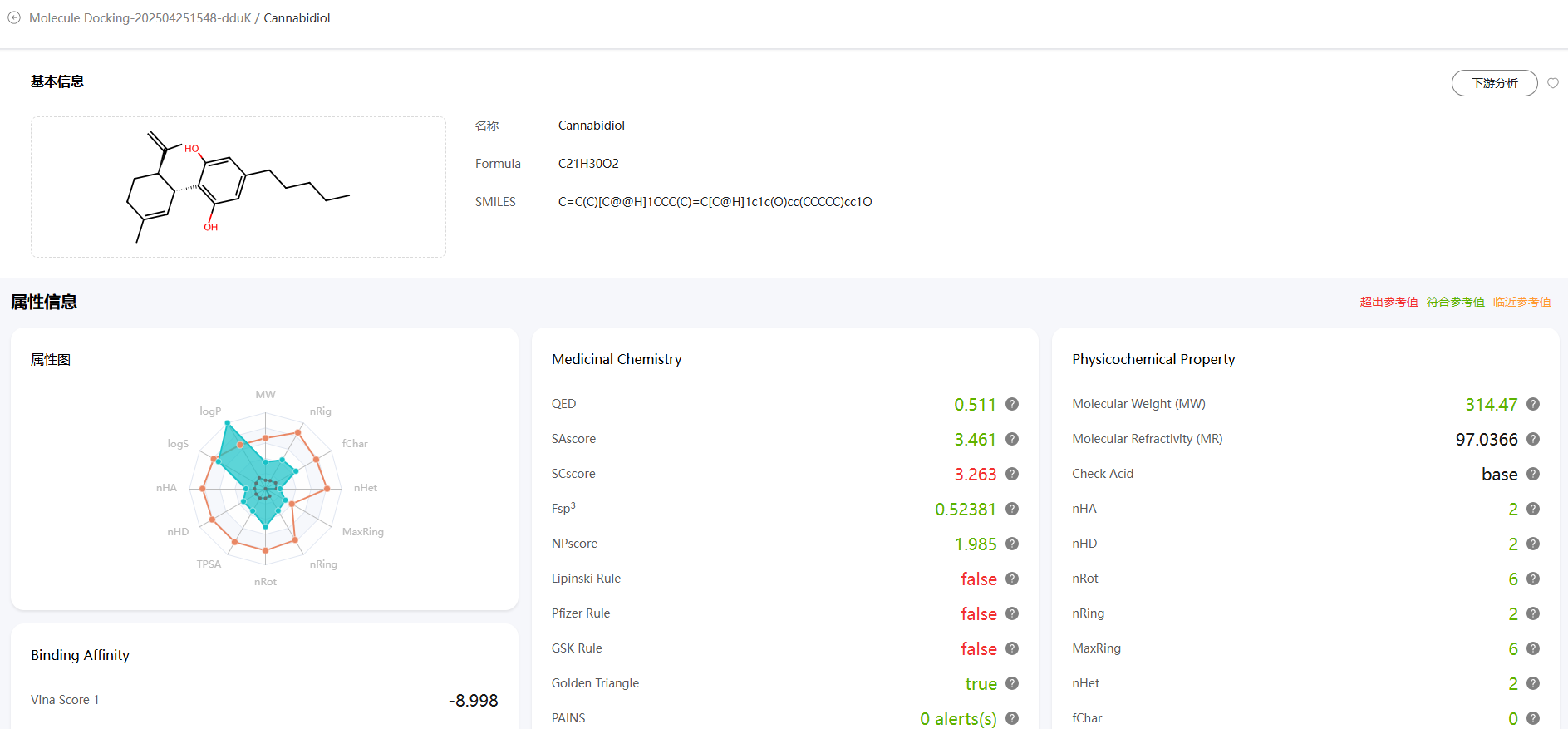
单击
 按钮,可以收藏对接结果,收藏后可直接在收藏夹页查看。
按钮,可以收藏对接结果,收藏后可直接在收藏夹页查看。 - 分子对接结果支持以卡片视图的形式进行查看,参考图14,在卡片视图中:
- 单击右上方的选择下拉框,可以选择分子的排序方式,将分子按照所选的排序方式进行展示。
图15 排序方式

- 单击每个分子卡片右上方的
 ,可以选择“查看详情”、“查看3D”、“下游分析”、“下载3D”。
,可以选择“查看详情”、“查看3D”、“下游分析”、“下载3D”。
- 查看详情:单击“查看详情”,跳转到分子详情页面。
- 查看3D:查看分子的3D视图。
- 下游分析:分子优化对应的下游分析为分子搜索、分子属性预测、分子优化和自由能微扰,单击“确定”即可创建。
- 下载3D:可以单击“下载3D”下载优化后的小分子或者复合物,小分子支持SDF和PDB格式,复合物只支持PDB格式。
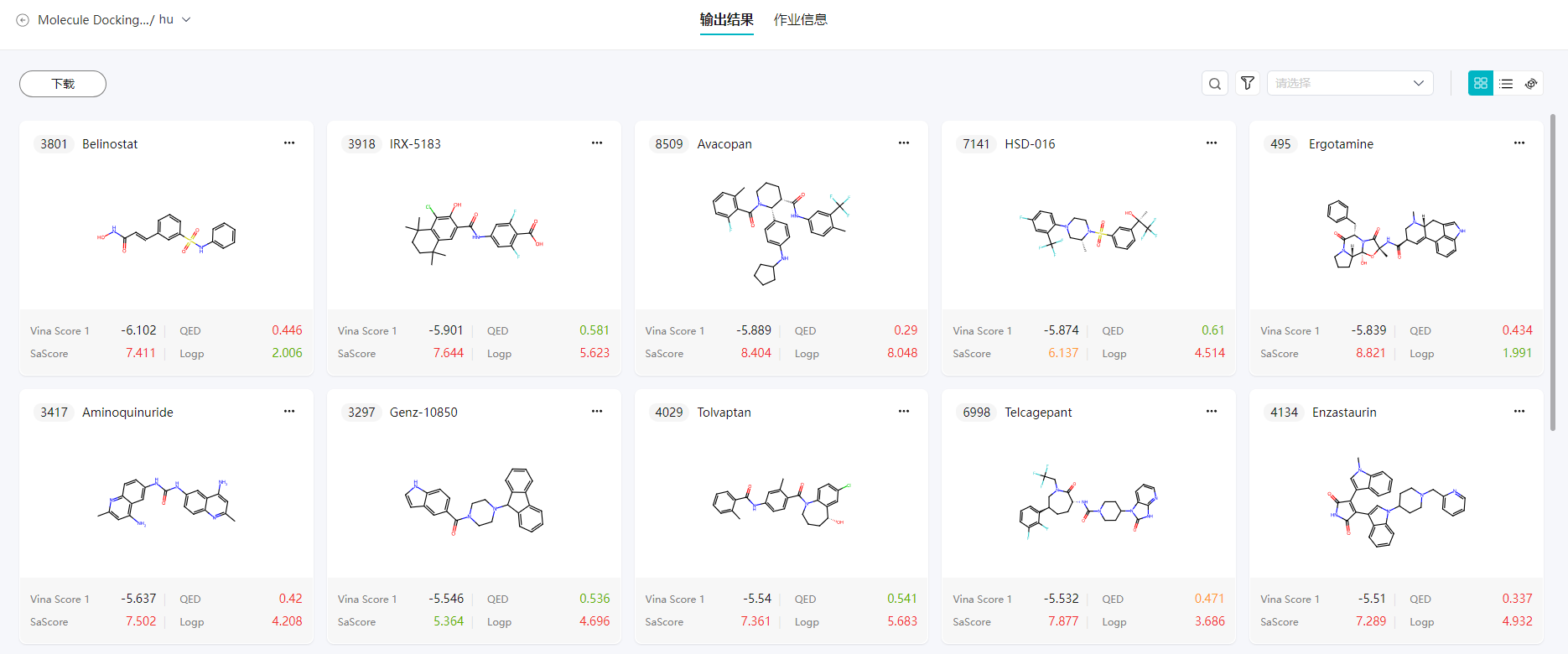
- 每个分子卡片上会展示相应分子序号与对应的参数Vina Score、QED、SaScore。
- Vina Score:代表将会计算对接结合能,并按照Vina Score进行排序。
- QED:代表分子的成药性。
- SaScore:代表合成可及性分数,旨在评估分子的合成难易程度。
- 单击右上方的选择下拉框,可以选择分子的排序方式,将分子按照所选的排序方式进行展示。
- 查看作业信息。
单击作业的“作业信息”页面可以查看作业的运行时间和配置参数。
图16 查看作业信息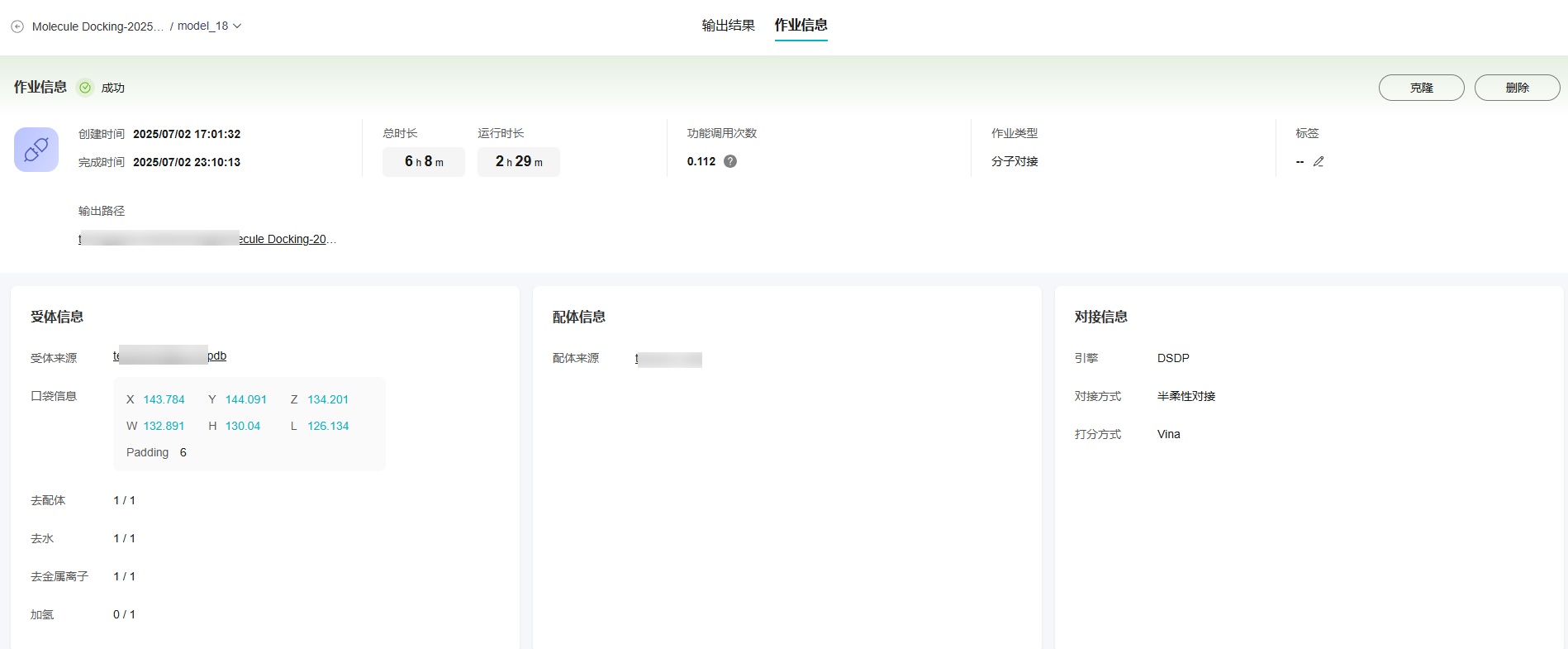
聚类分析
目前分子对接返回的结果小分子数较多,无法进行批量分析,通过一些聚类的辅助方式能更好地选择分子。从每个类里挑选出一两个分子进行后续分析和验证,提高分析的效率和分析质量。也可以通过聚类找出一些关键的骨架,来进行下游的分子优化和分子属性预测等。
- 在输出结果页面左上角单击“聚类分析”后,系统开始进行分析,同时显示“聚类分析中”。
图17 聚类分析
- 待聚类分析完成后,单击“查看聚类结果”。进入聚类结果页。
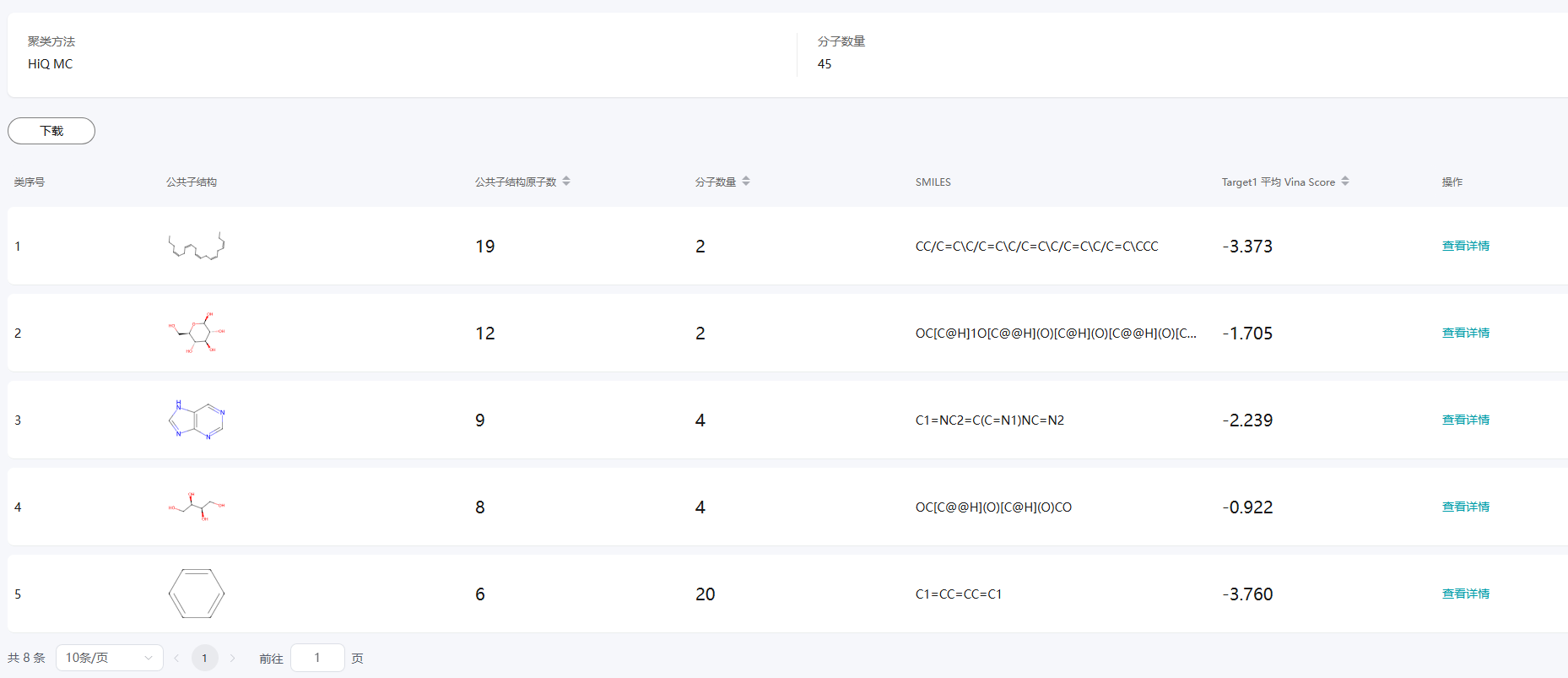
- 在聚类结果页面,可以查看每个聚类的分子数量等信息。
图18 查看聚类结果
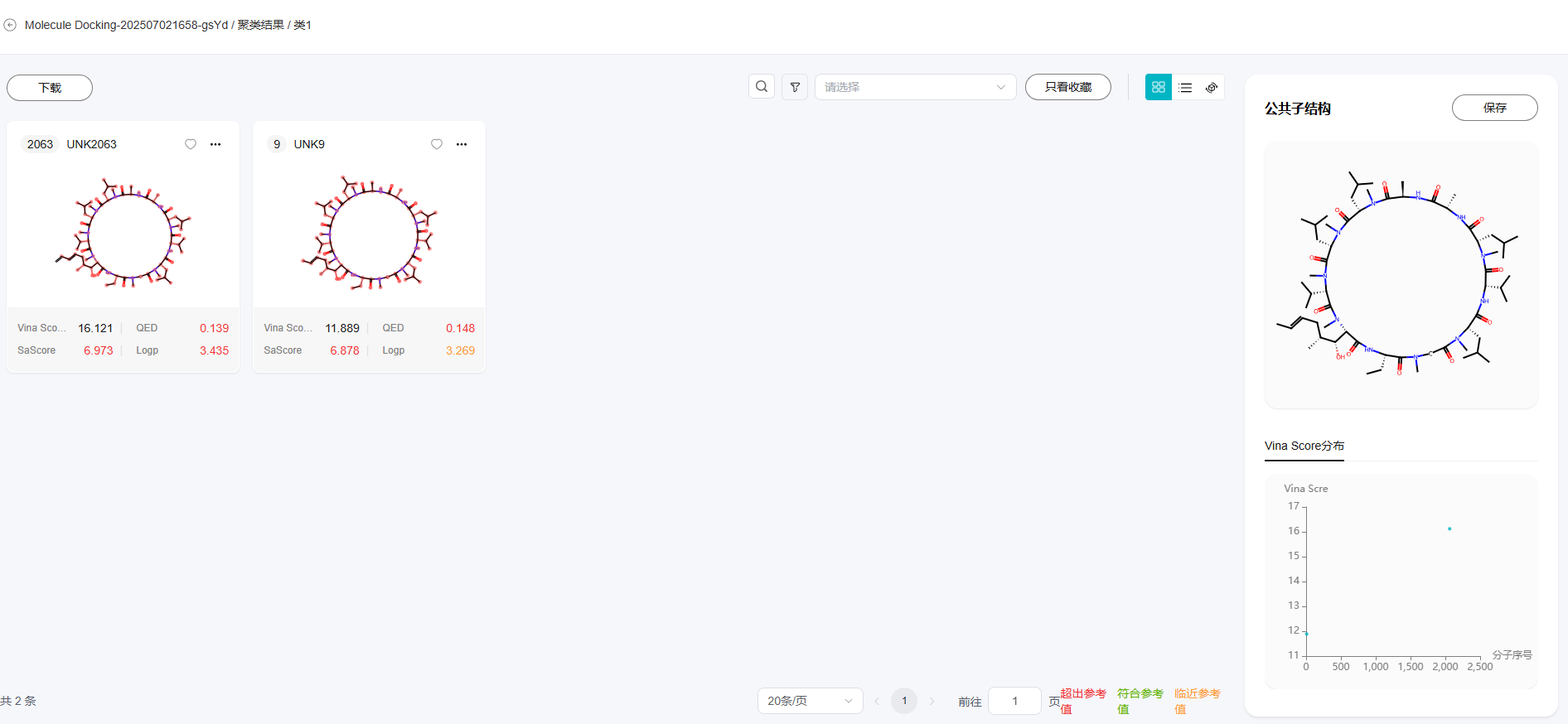
- 单击某个聚类的操作列的“查看详情”,即可进入聚类详情页面,聚类详情页支持以卡片、列表以及3D的形式查看。默认展示卡片页面,用户可自行进行切换。
图19 查看聚类详情

- 每个结果页面只用进行一次聚类分析操作。
- 聚类结果是存成文件,如果文件被删除或者获取不到,会有警告“聚类结果不存在” 。此时可以单击“重新聚类分析”。
- 如果聚类失败,根据提示失败原因解决问题后,可单击“重新聚类分析”。
相关参数
分子对接的具体参数,请参考小分子药物设计相关参数。