操作流程
自定义镜像运行流程的操作流程如表1所示。
|
操作步骤 |
说明 |
|---|---|
|
购买并绑定计算集群。 |
|
|
在AI科学计算平台创建新空间,并在空间范围内上传数据、创建作业等。 |
|
|
从dockerhub下载镜像。 |
|
|
上传bioifo_demo镜像至AI科学计算平台。 |
|
|
基于bioifo_demo镜像创建multifasta应用。 |
|
|
基于multifasta应用搭建流程并启动作业。 |
|
|
查看作业信息、输入输出数据等信息。 |
步骤一:绑定资源
AI科学计算服务的分析作业是以容器的形式在CCE集群中运行,用户可在平台绑定本账号下的CCE集群用于此功能的运行。

- 当前支持CCE集群1.28和1.31版本。
- 如果您没有可用的CCE集群,需先创建CCE集群。CCE 1.28仅支持使用api接口创建,CCE 1.31支持通过页面创建。
- 如果您已有CCE集群,但CCE集群版本低于1.28,则可参考升级集群的流程和方法,建议将集群升级至1.28或1.31版本。
具体操作步骤如下:
- 购买CCE集群并进行相关配置
首先,用户需要购买CCE集群并进行相关配置,以满足AI科学计算服务使用的要求,具体操作指导如下:
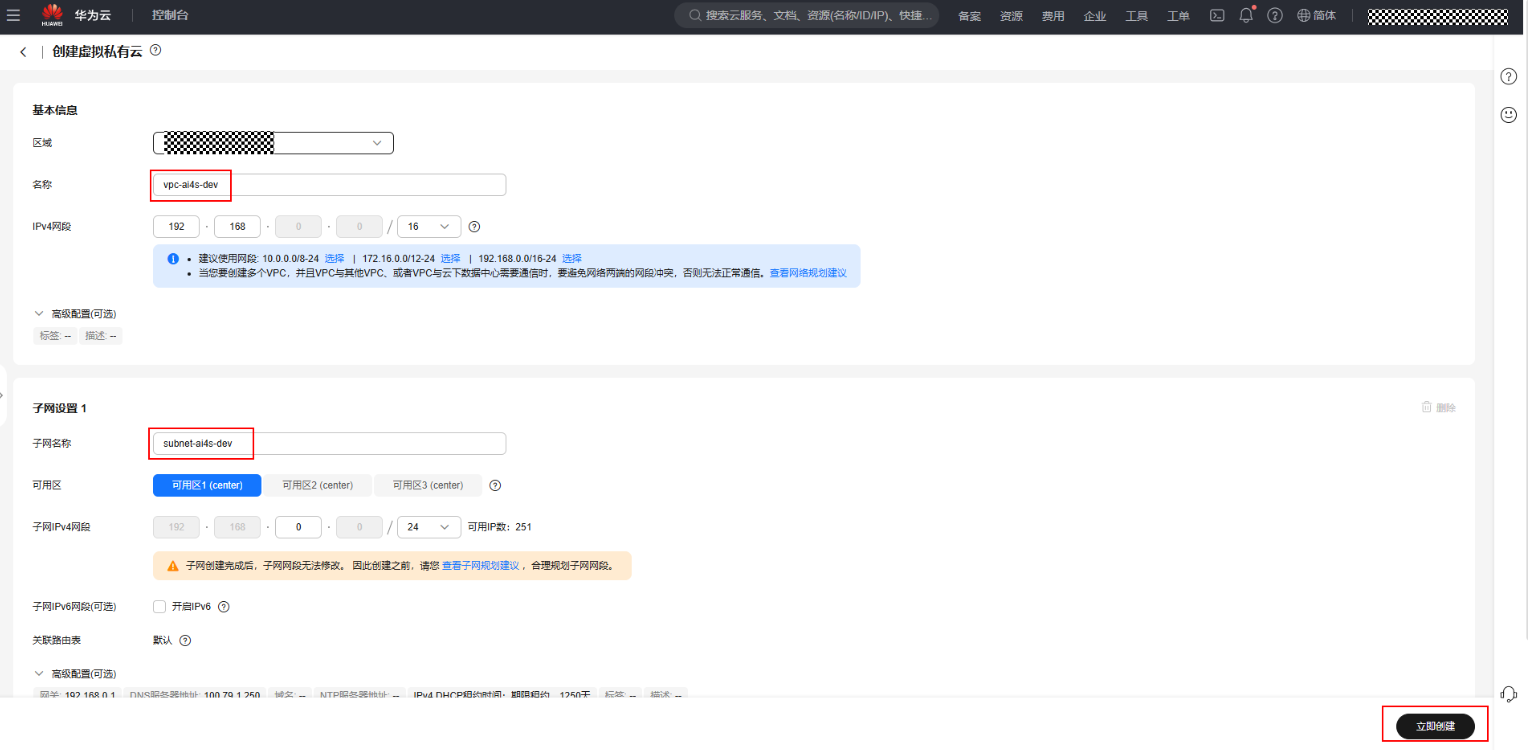
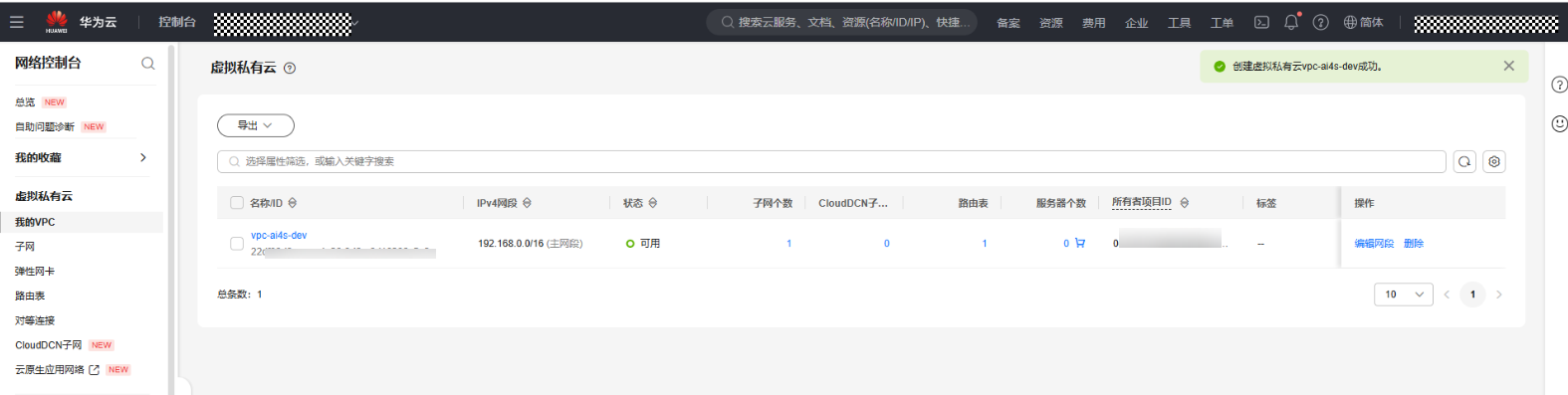
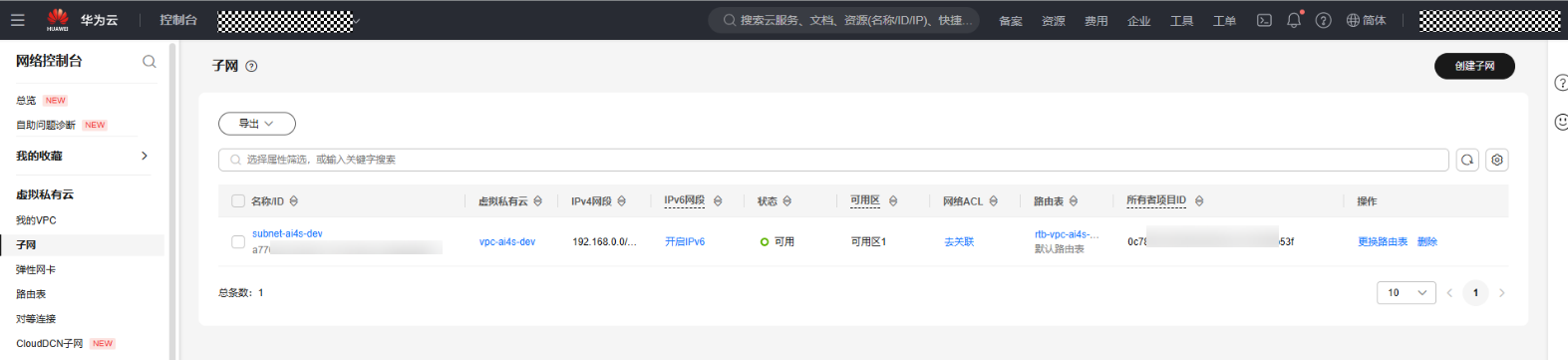
- 创建虚拟私有云
- 创建CCE集群
- 左上角打开服务搜索页面,搜索“cce”,单击进入“云容器引擎”服务。
图7 云容器引擎
- 新账号首次进入“云容器引擎”服务界面,会弹窗提示授权,单击“确定”按钮即可。
如果没有弹窗,说明以前创建过授权,可以跳过此步骤继续往下进行。
图8 提示授权
- 单击右上角“购买集群”按钮。
图9 购买集群
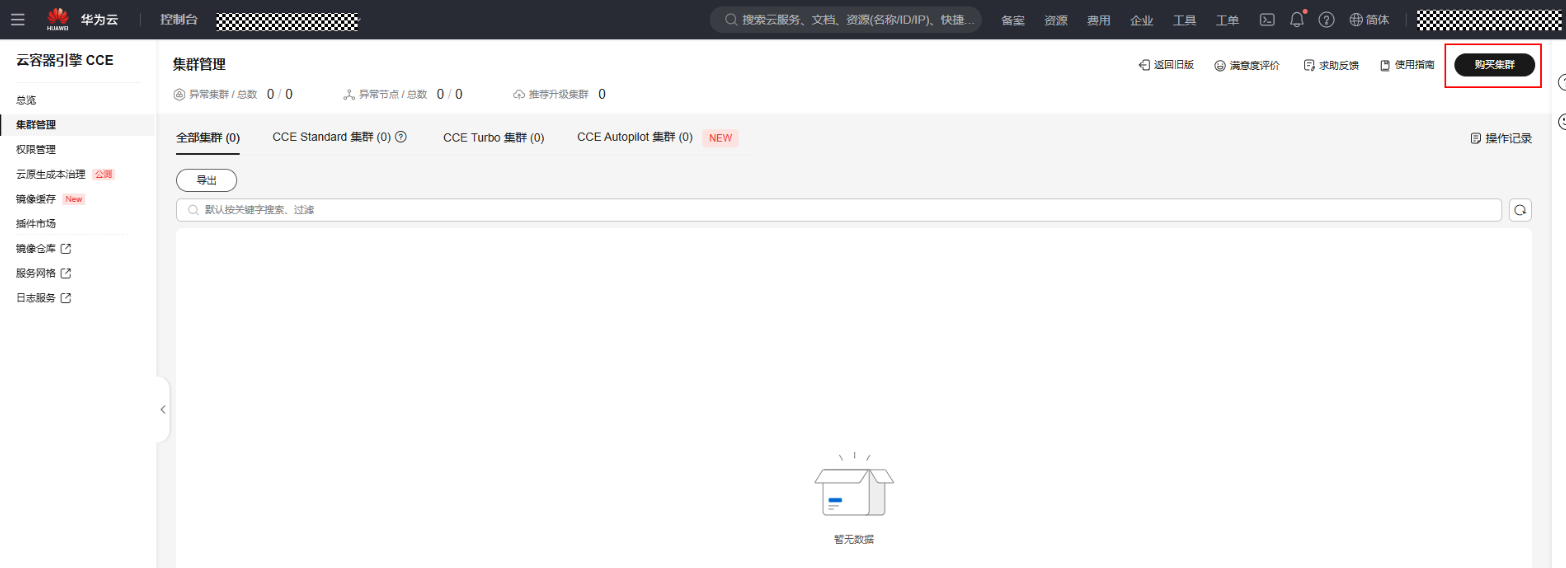
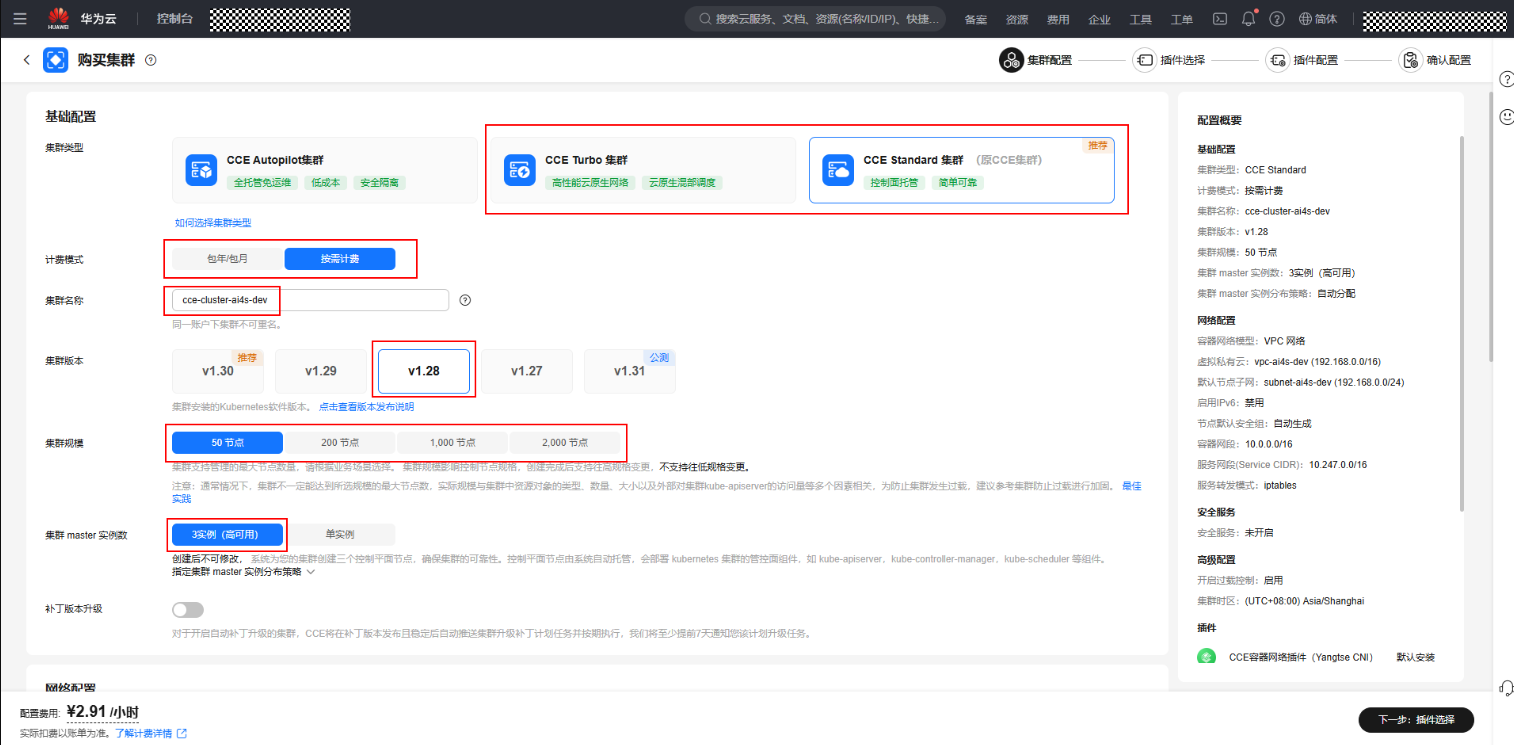
- 在集群配置页面,根据自己需要进行设置,基础配置可参考如下:
集群类型:可选“CCE Turbo 集群”或“CCE Standard 集群”。
计费模式:可选“包年/包月”或“按需计费”。
集群版本:建议选择v1.31。
集群规模:根据使用场景可选“50节点”、“200节点”、“1000节点”或“2000节点”。
集群master实例数:建议选择“3实例(高可用)”。
- 网络配置可参考如下:
虚拟私有云与默认节点子网:选择上一步骤中创建的虚拟私有云与子网。
容器网络模型:建议选择“容器隧道网络”。
网络策略按钮:默认打开。
其他配置保持默认。
最后单击右下角“下一步:插件选择”按钮。图11 网络配置-CCE Standard 集群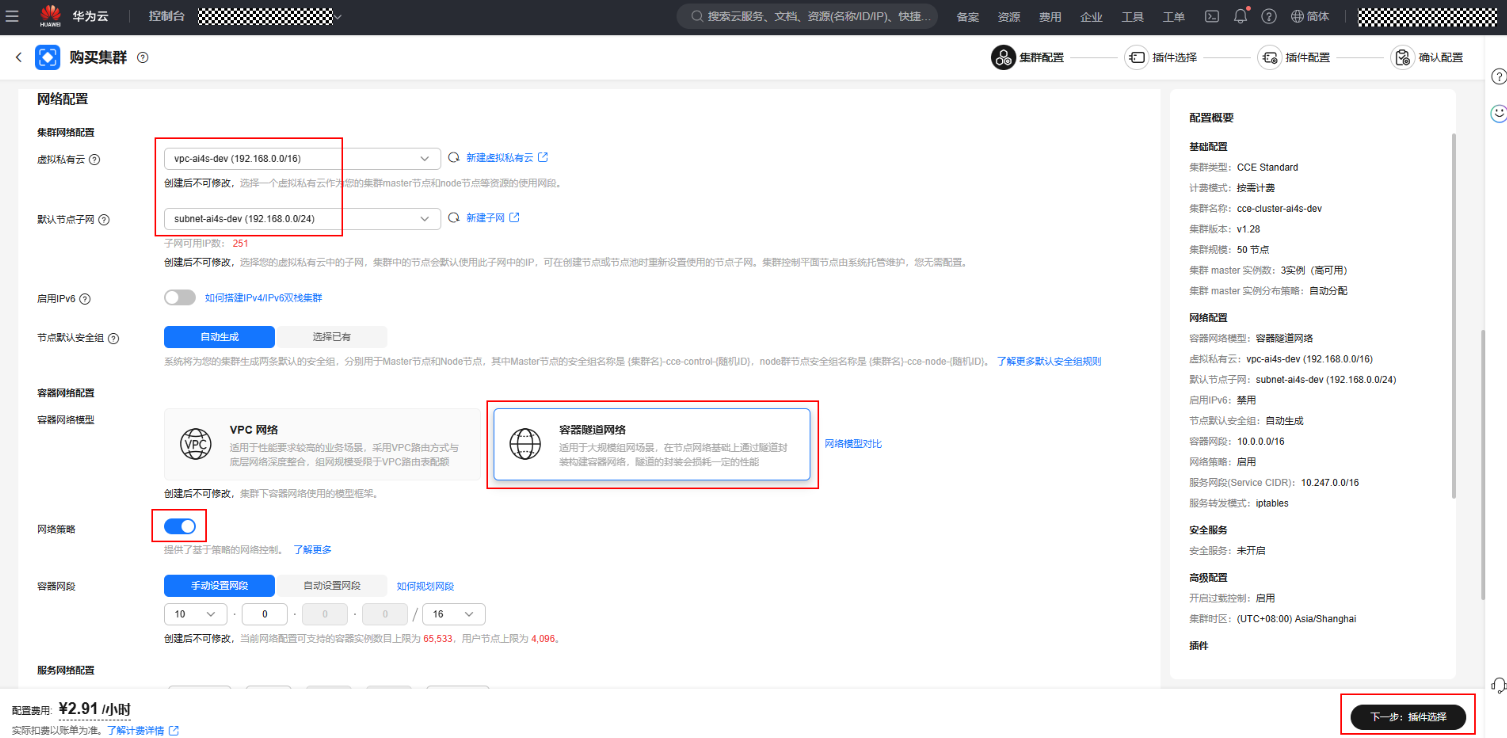
文档以“CCE Standard 集群”为例,如果选择创建“CCE Turbo 集群”,则容器网络模型只有“云原生网络2.0”,其他配置方式相同。
图12 网络配置-CCE Turbo 集群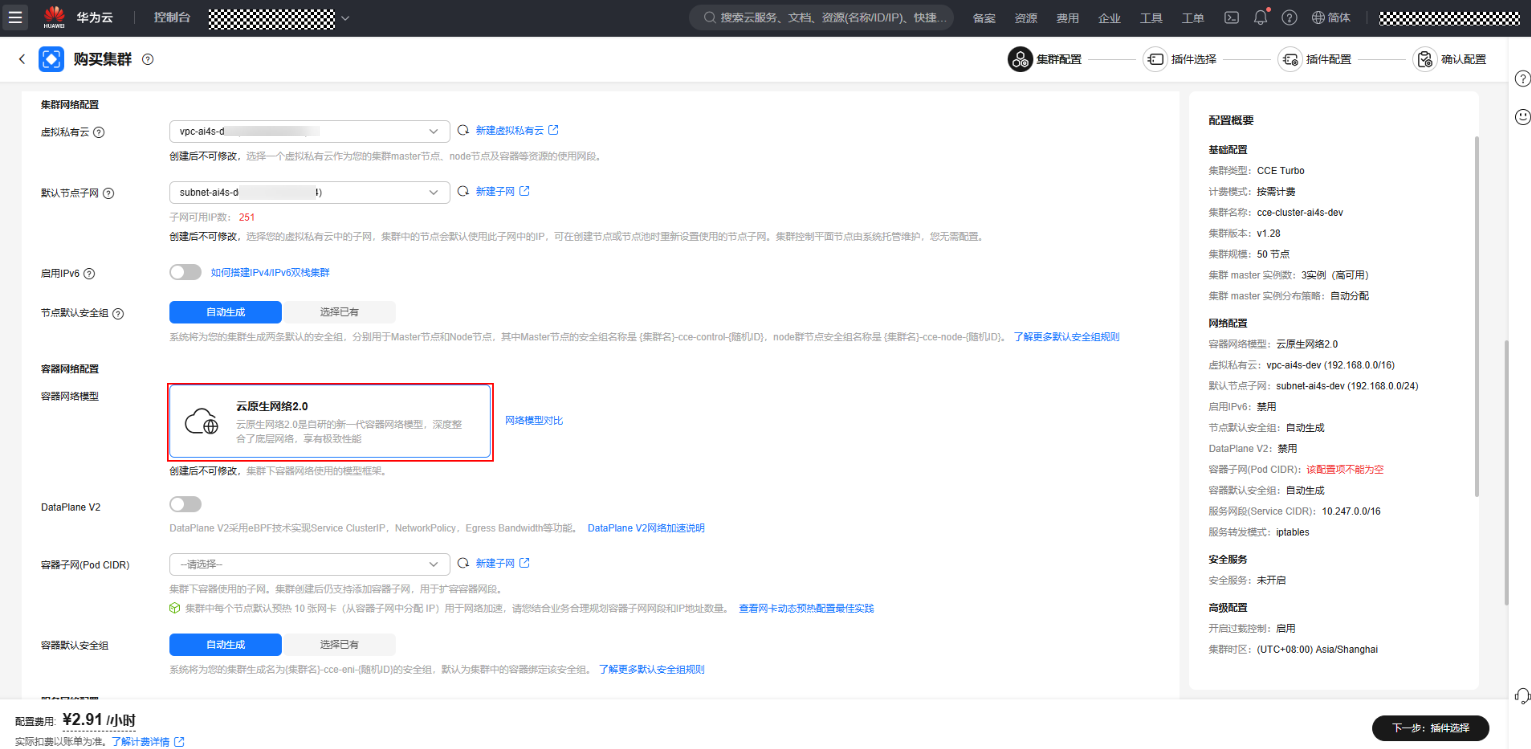
- 插件选择页面,单击勾选“Volcano调度器”、“云原生日志采集插件”、“CCE节点故障检测”插件,单击右下角“下一步:插件配置”按钮。
图13 插件选择
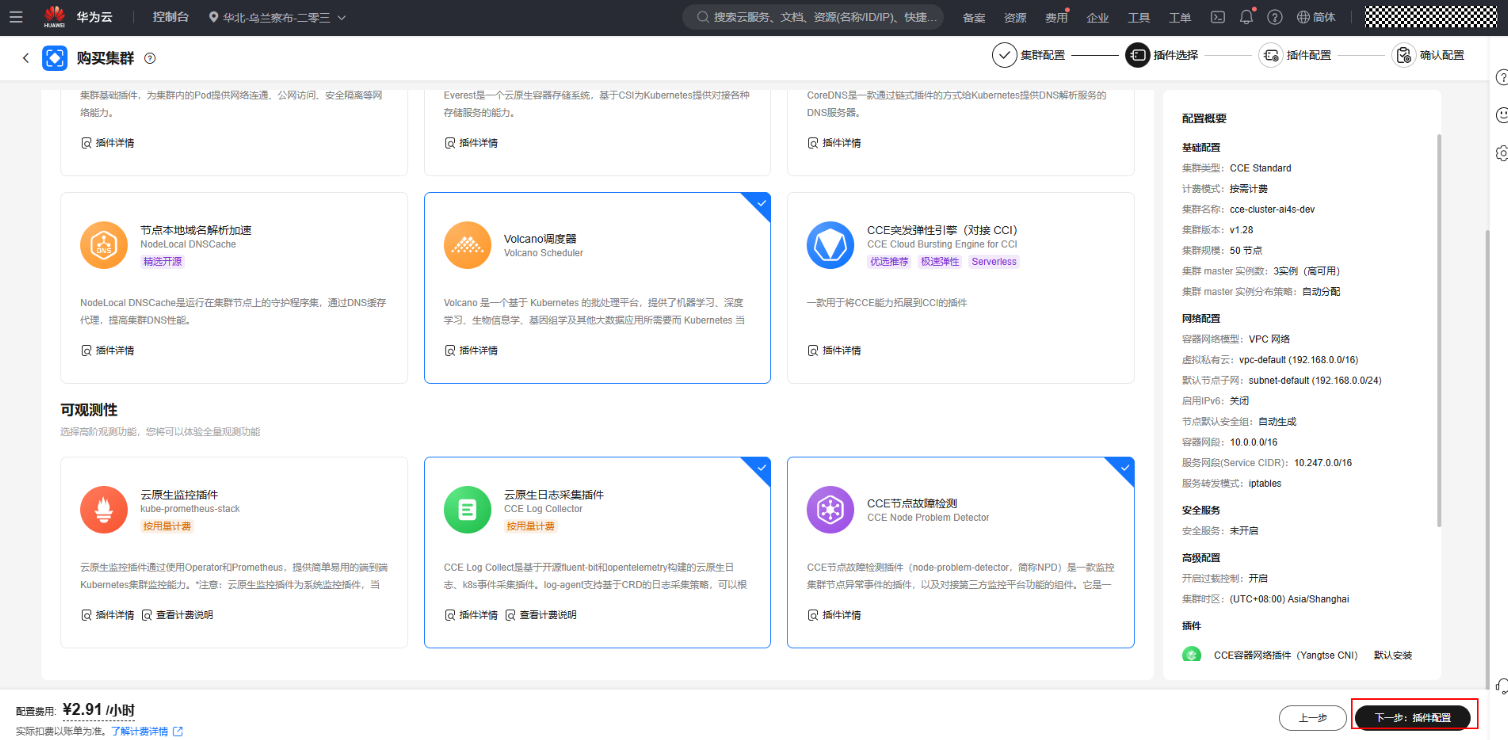
- 插件配置页面,不需要操作,直接单击右下角“下一步:确认配置”按钮。
图14 插件配置
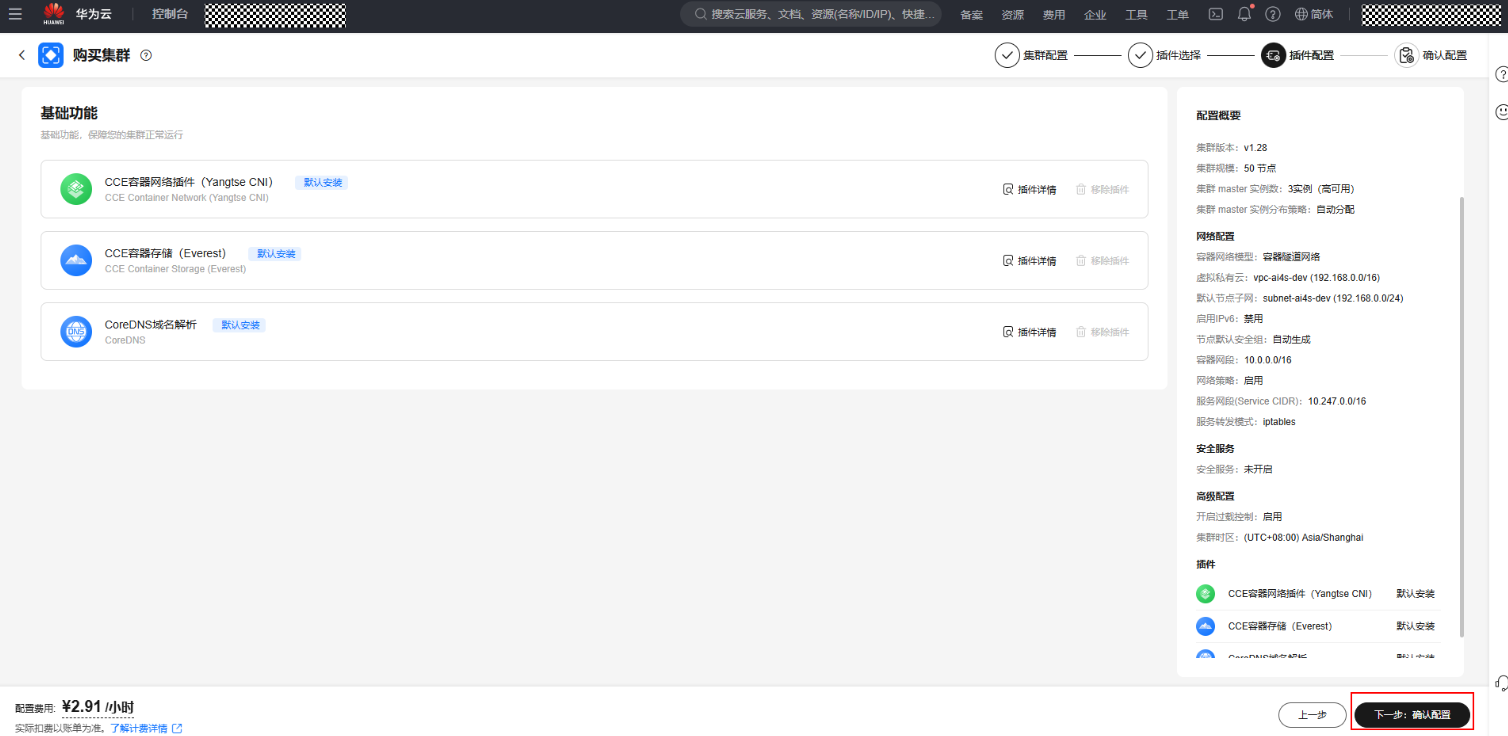
- 确认配置页面,左下角勾选“我已阅读并知晓上述使用说明”,然后单击右下角“提交”按钮。
图15 确认配置
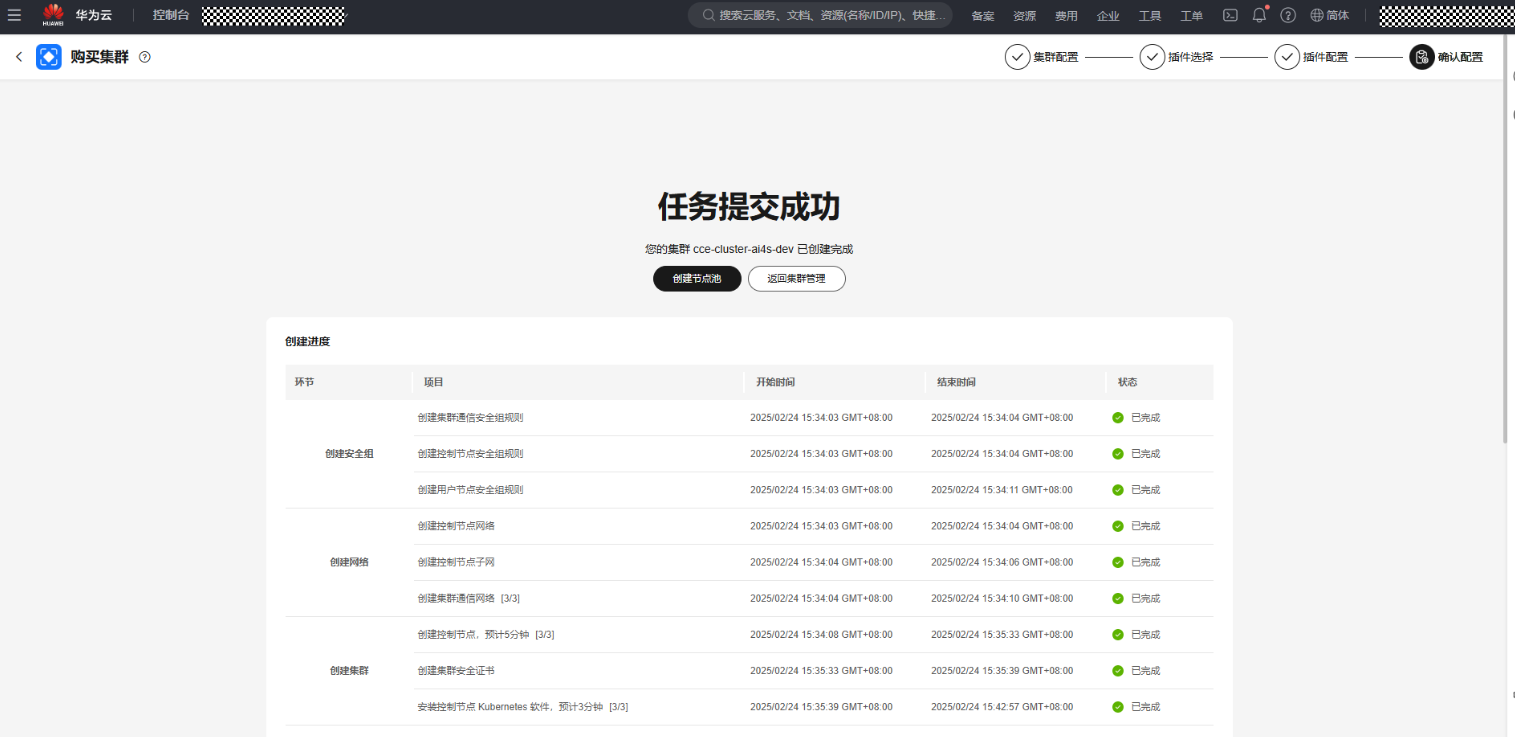
- 创建集群任务提交成功后,等待几分钟,集群创建成功,页面显示如图16 任务提交成功。
- 云容器引擎集群管理页面,集群展示如图17 集群管理所示。
- 左上角打开服务搜索页面,搜索“cce”,单击进入“云容器引擎”服务。
- 打开日志采集配置
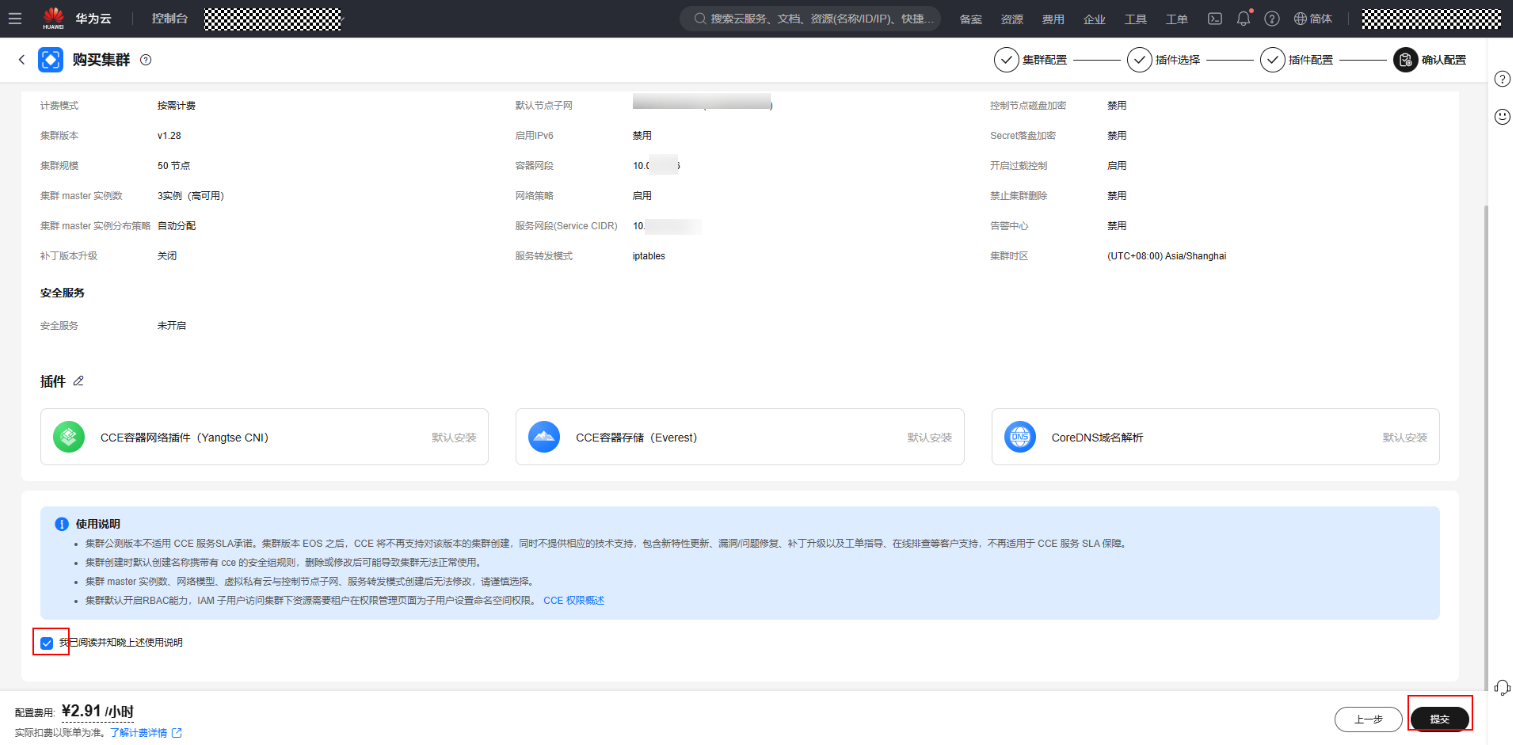
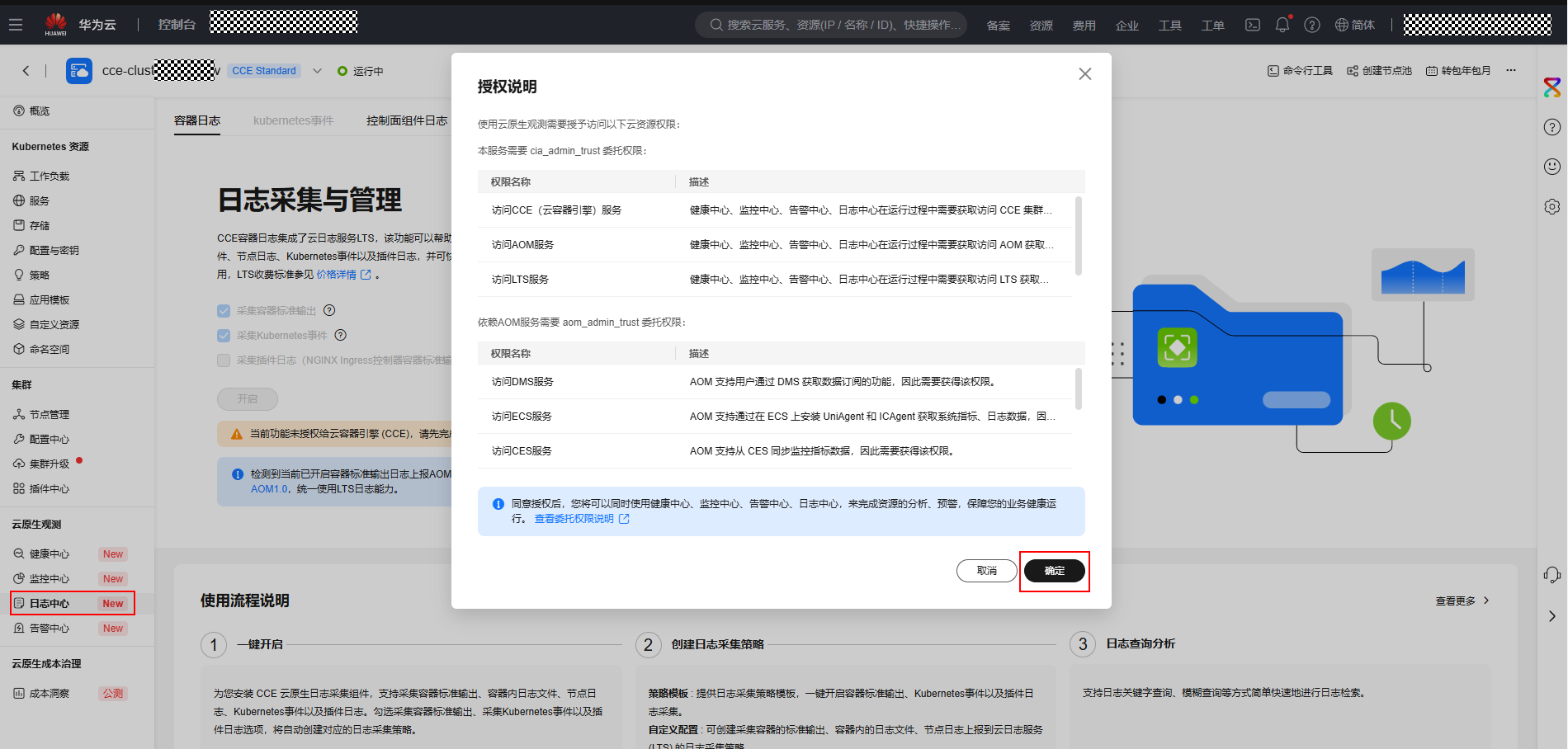
- 单击集群名称,找到日志中心-->授权说明弹窗,单击“确定”按钮。
图18 授权说明
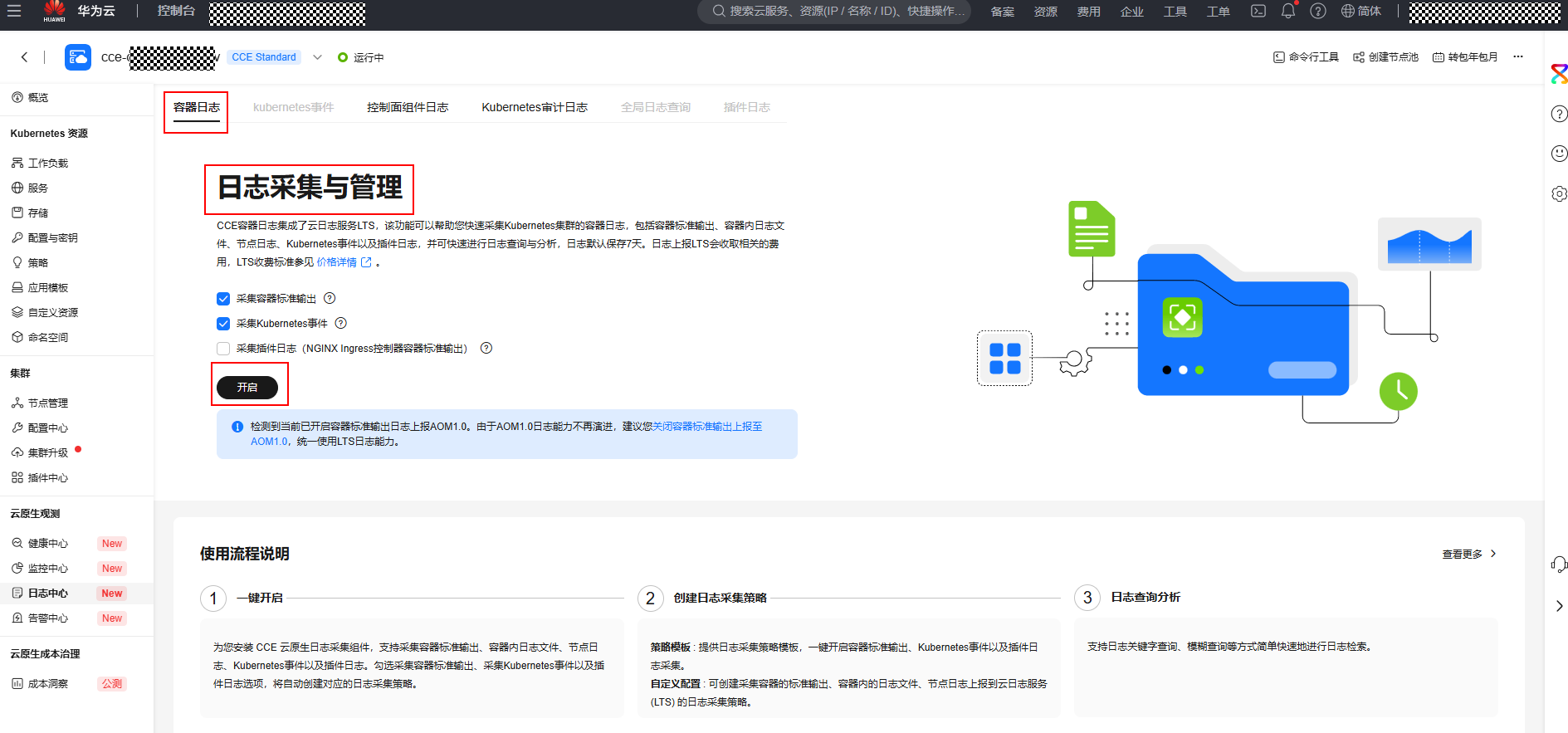
- 在容器日志界面,日志采集与管理下方,单击“开启”按钮。
图19 开启日志采集与管理
- 单击集群名称,找到日志中心-->授权说明弹窗,单击“确定”按钮。
- 节点标签配置
- 用户根据自己需要创建好节点后,单击右侧“更多 > 标签管理”按钮。
图20 标签管理
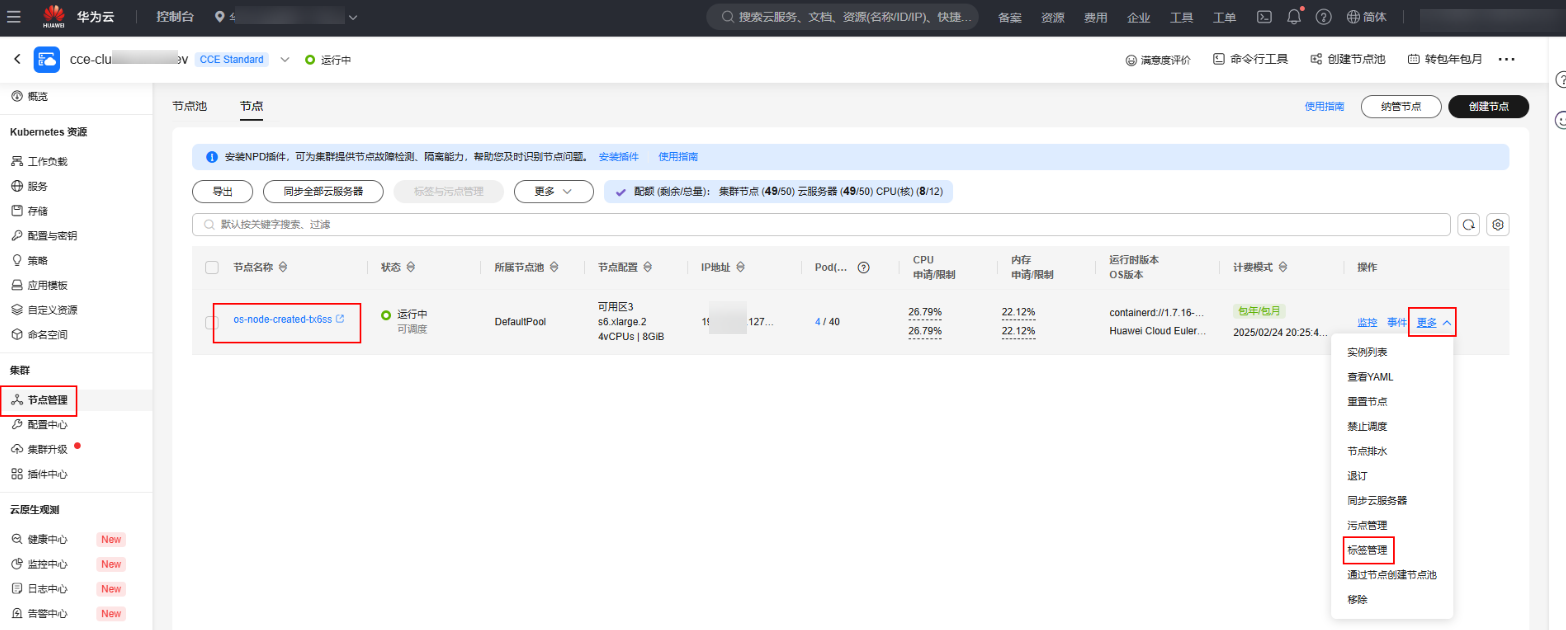
- 在标签管理弹窗单击“+添加”按钮添加标签,然后单击“确定”按钮。
图21 添加标签
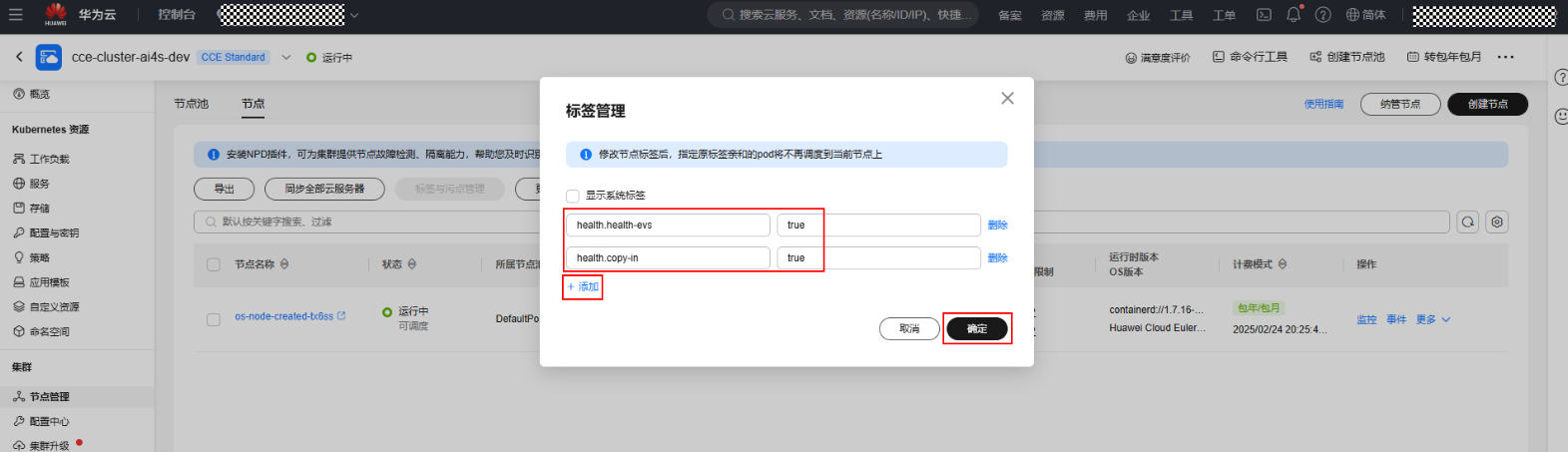
平台功能涉及标签及对应功能如表2所示,请用户根据需求进行标签配置,以控制分析作业的部分任务的调度节点。
表2 平台功能涉及标签及对应功能 标签
功能
health-evs=true
若存在节点挂载了容量较大的数据盘,分析作业的加速方式选择了“本地盘加速”,则需在该节点配置标签,使得作业可调度到该节点上。
health.copy-in=true
若分析作业的加速方式选择了“IO加速”,则需选择节点配置标签使得作业可调度到该节点上。
health.{自定义}=true
若分析作业的高级参数中配置了“计算节点标签”,则需在希望调度上的节点配置相应标签。
若用户新增了其他节点,需重复以上动作进行标签配置。
- 用户根据自己需要创建好节点后,单击右侧“更多 > 标签管理”按钮。
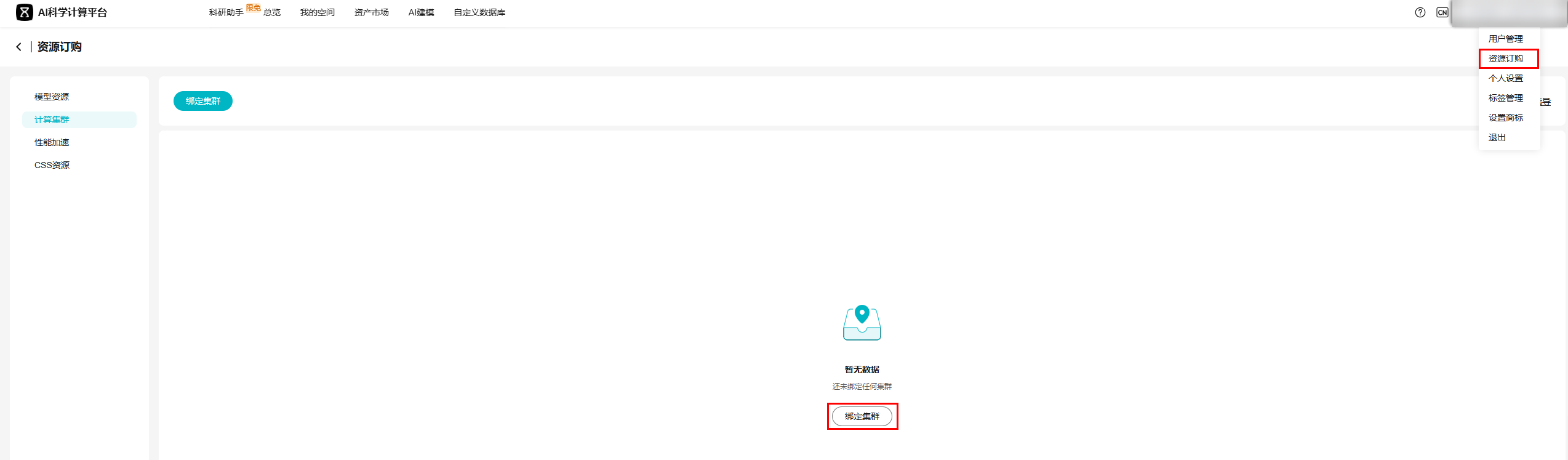
- 完成CCE集群创建后,用户可进入AI科学计算平台进行计算集群绑定。
- 单击右上角账号名,选择“资源订购”,进入“计算集群”页面,单击“绑定集群”按钮。
图22 绑定集群
- 在“绑定集群”弹窗中,选择上面步骤创建的CCE集群。
图23 绑定集群2
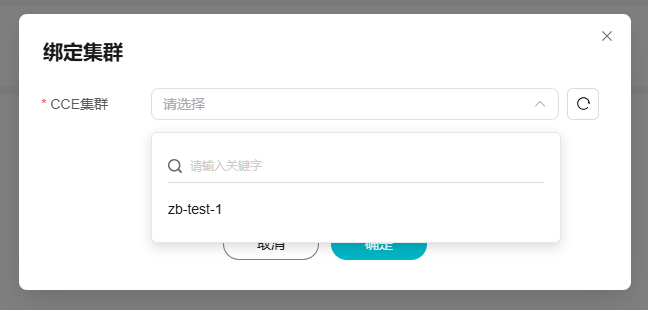

此处仅展示符合AI科学计算服务约束的CCE集群。若未展示,请检查集群类型、集群版本是否在支持的版本范围内,集群状态是否为可用状态。
- 单击绑定后,查看集群绑定状态为“绑定中”,需等待几分钟。
图24 绑定中
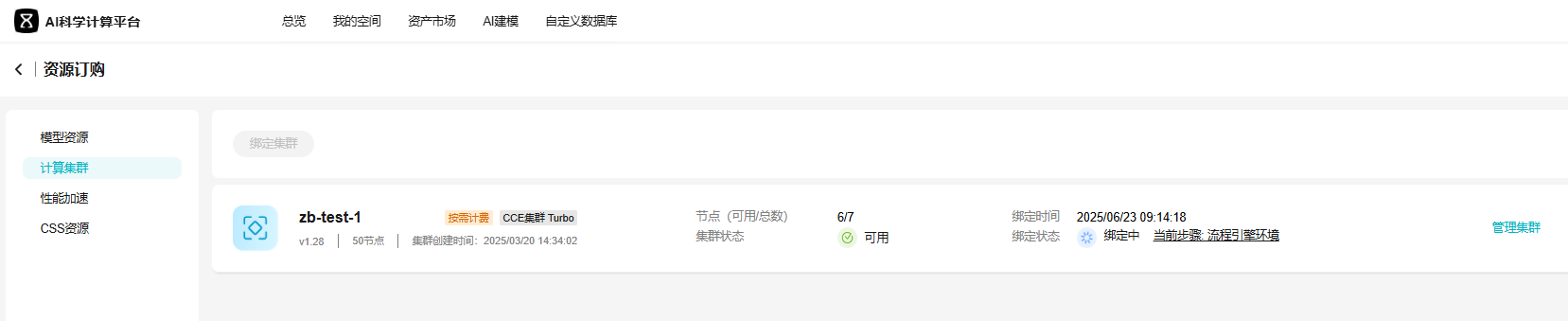
- 单击右上角账号名,选择“资源订购”,进入“计算集群”页面,单击“绑定集群”按钮。
步骤二:创建空间
您可以在AI科学计算平台创建新空间,并在空间范围内上传数据、创建作业等。“空间列表”展示了当前用户有权限访问的空间。
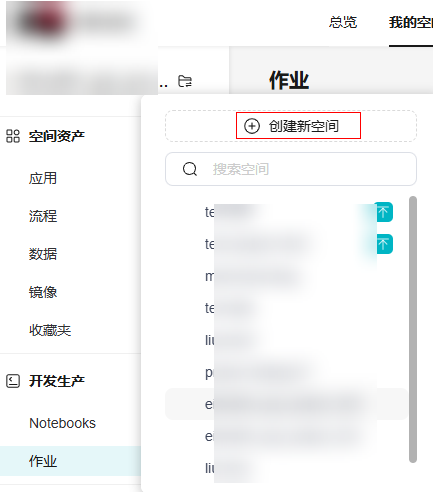
创建新空间时可直接创建一个OBS桶,也可使用已有的桶。
创建桶的命名遵循obs桶命名规则:
- 需全局唯一,不能与已有的任何桶名称重复,包括其他用户创建的桶。用户删除桶后,立即创建同名桶或并行文件系统会创建失败,需要等待30分钟才能创建。
- 长度范围为3到63个字符,支持小写字母、数字、中划线(-)、英文句号(.)。
- 禁止两个英文句号(.)相邻,禁止英文句号(.)和中划线(-)相邻,禁止以英文句号(.)和中划线(-)开头或结尾。
- 禁止使用IP地址。
步骤三:从dockerhub下载镜像
- 在linux环境上配置docker环境。
curl -fsSL get.docker.com -o get-docker.sh sh get-docker.sh
- 配置完成后,运行docker命令,可查看docker的信息。
图27 docker信息
- 构建镜像。
- 可以在dockerhub官网上进行搜索,选择出对应的名称与版本。
- 可以使用docker search命令进行搜索镜像。
- 若dockerhub上没有想要的镜像,可以利用基础的操作系统镜像进行构造,构造方式请参见用户指南。
- 选择和获取镜像。
搜索到的镜像列表中选择一个镜像进行pull,这里以pegi3s/fastqc为例。
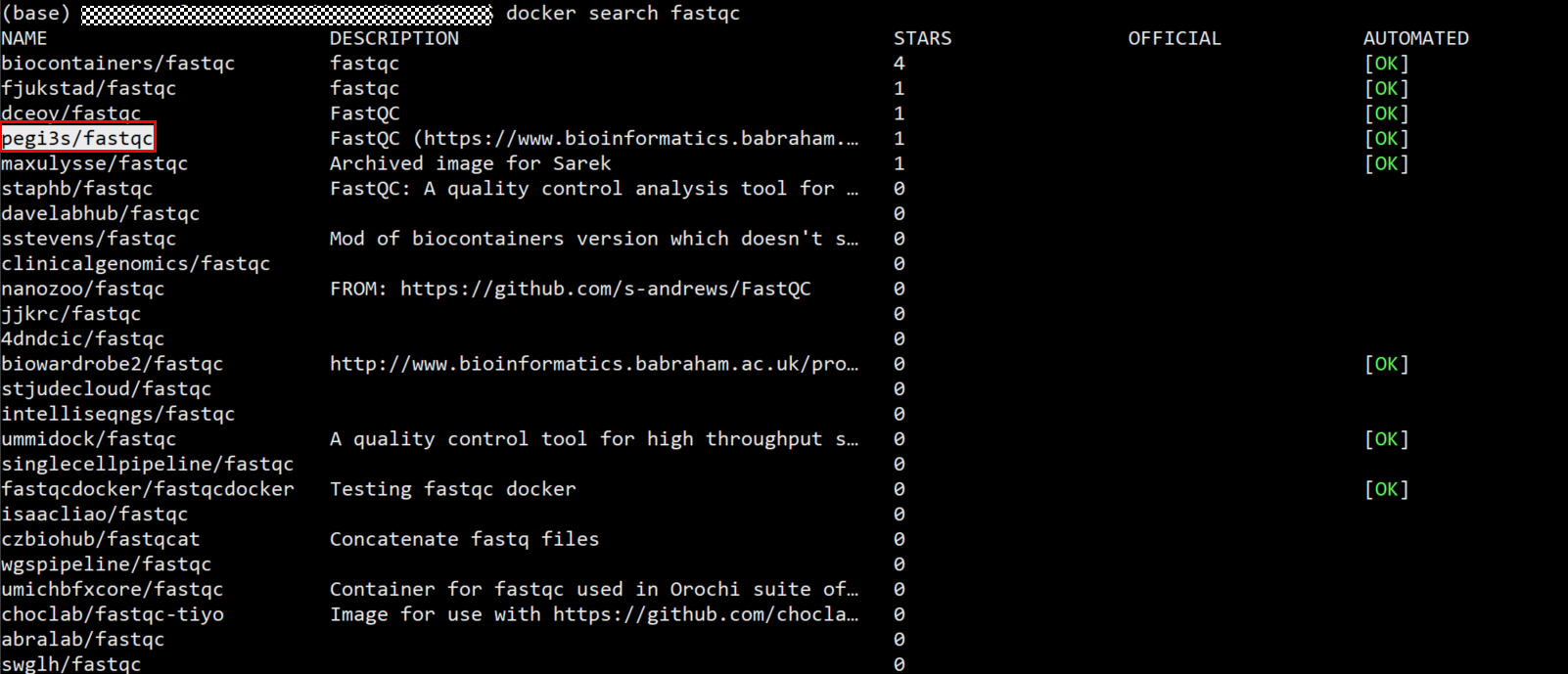
- pull镜像。
若该镜像没有lastest版本则需进入dockerhub官网进行查询。

- 查看镜像是否pull成功。
用docker images命令查看镜像是否pull成功。如果您本地已经有很多镜像,防止查询出全部镜像,可在docker images命令后面添加 | grep pegi3s使查询更精准。

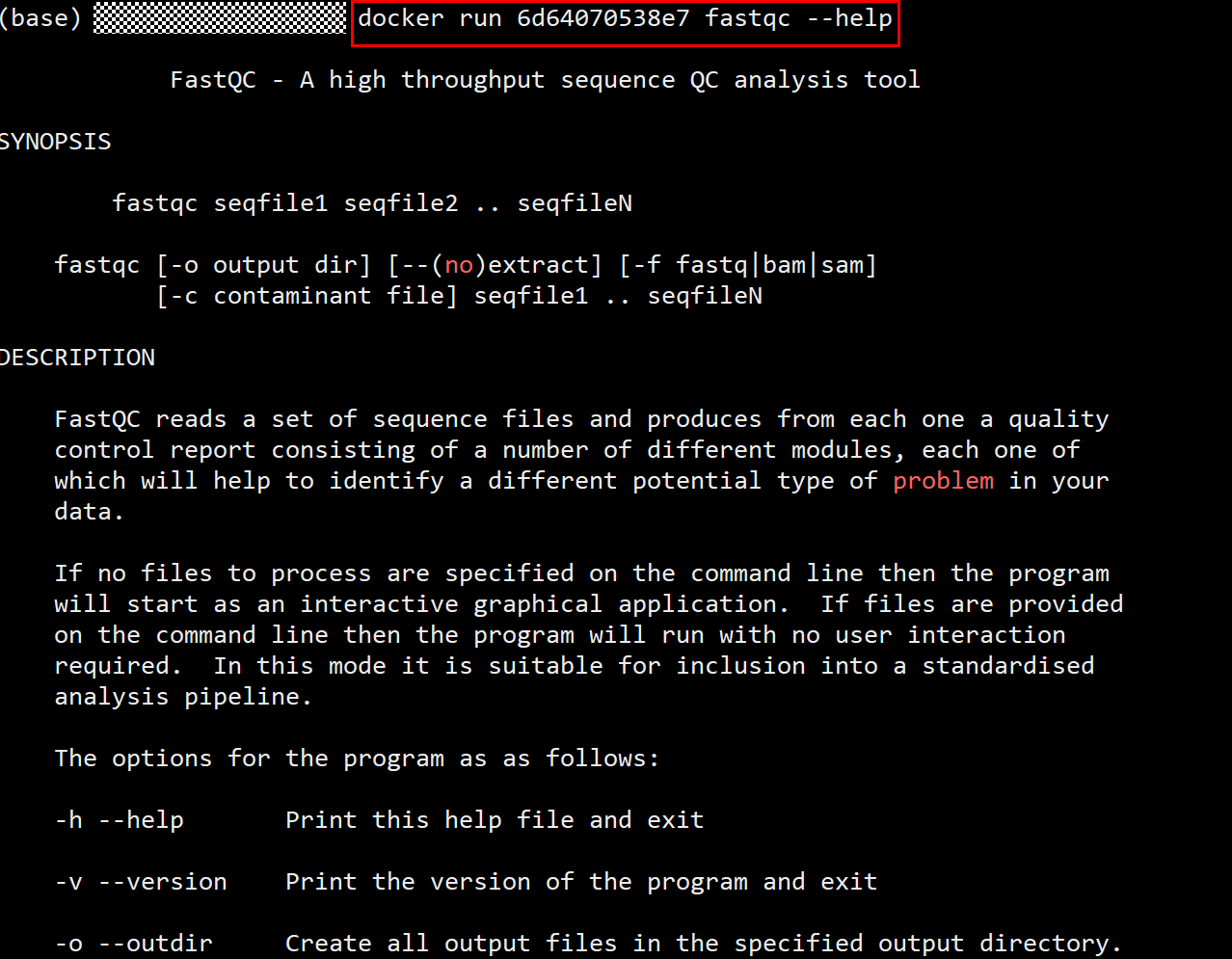
- 查询软件命令,以fastqc为例。熟悉该软件时可跳过。
复制上一步查找到的fastqc的id。利用docker run imagesId fastqc--help命令进行查询。fastqc--help可以替换成任何fastqc的命令,与linux类似。
步骤四:上传镜像至AI科学计算平台
- 配置命令行工具。
若已完成配置,可跳过此步骤。
- 在命令行工具所在的目录,使用switch命令进入AI科学计算平台的空间中。
# 命令结构 ./ai4s switch project <project-name> # 命令示例,例如进入到名为user-tutorials的空间中 ./ai4s switch project user-tutorials
- 使用docker tag命令给要上传的镜像打标签。
# 给上传的镜像打标签 ./ai4s docker tag bioinfo_ubuntu2204 bioinfo_demo:latest

- 使用docker push命令将镜像上传至AI科学计算平台。
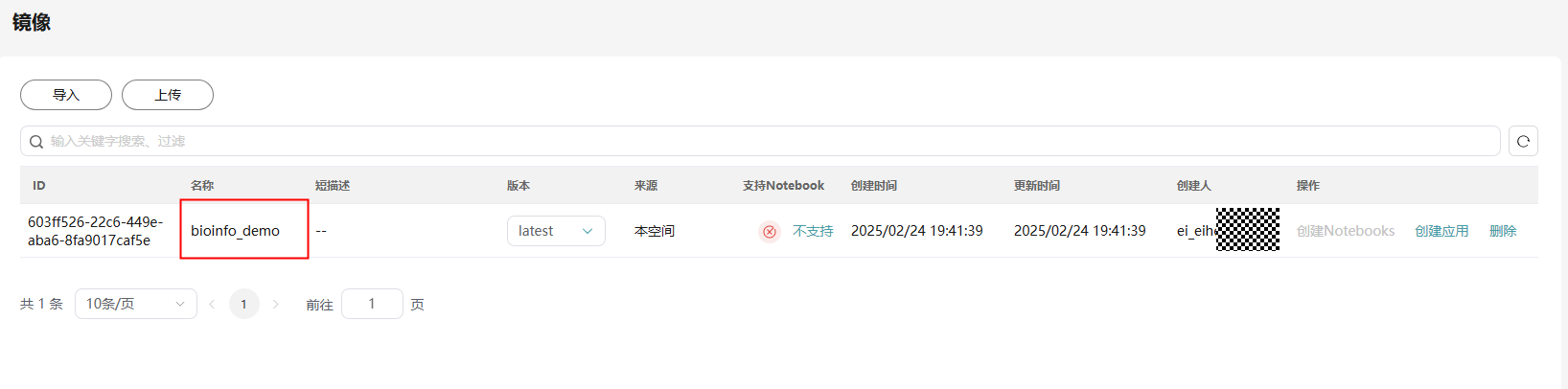
# –t APP 可不加,在平台上也可以对其进行设置,push前需要先switch到对应的空间中 ./ai4s docker push bioinfo_demo:latest –t APP

上传成功后,可以转至平台查看已经上传的镜像。
步骤五:创建multifasta应用
上一步骤中,已经将bioifo_demo的镜像上传至平台,本步骤使用该镜像创建multifasta应用。
- 单击“创建应用”,进入创建应用页面。
图28 创建应用
- 填写应用的基本信息,包括“名称”、“版本”、“图标”、“标签”、“短描述”和“长描述”。
图29 基本信息
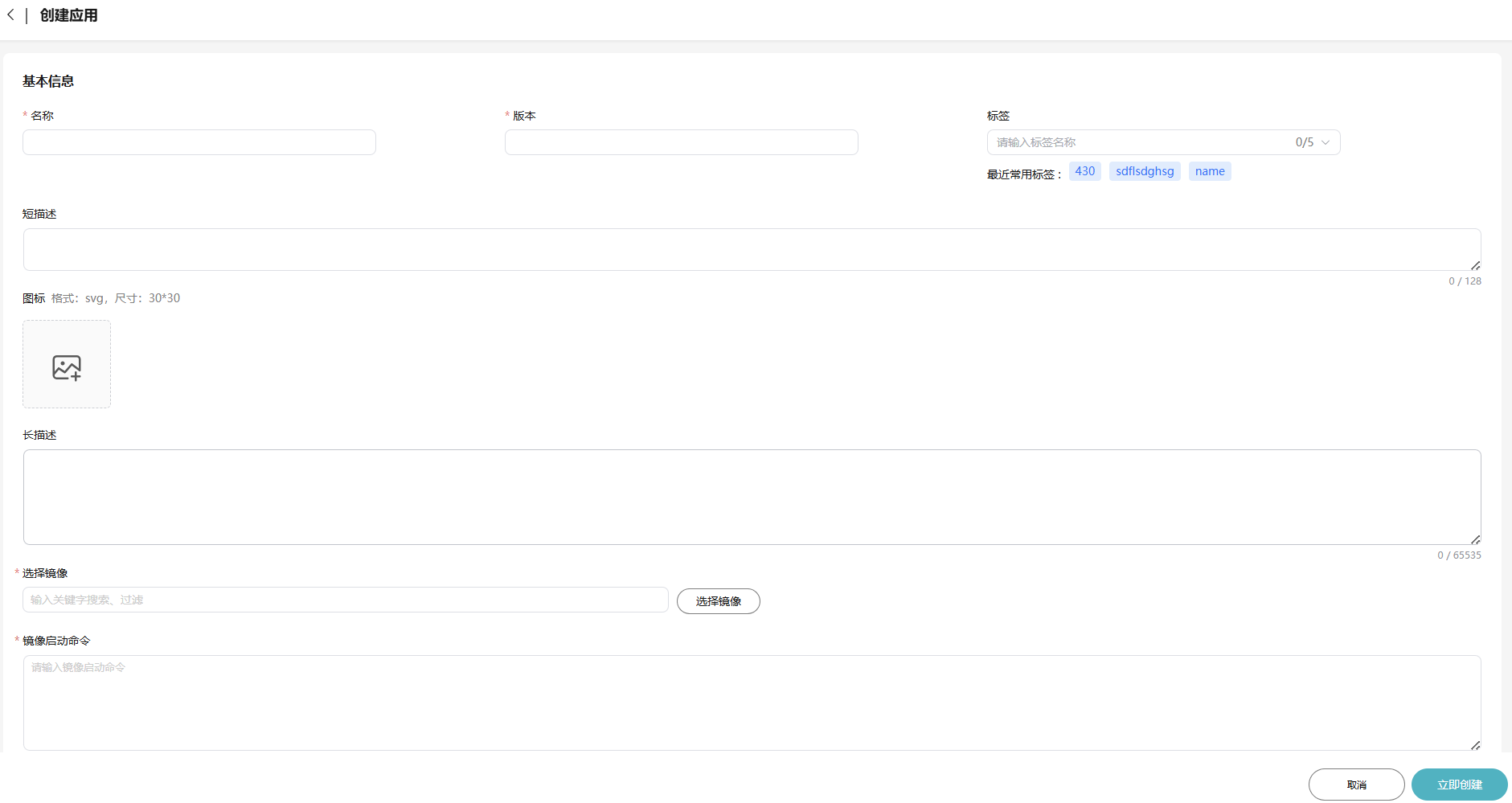
- 选择镜像和镜像版本。
- 填写镜像启动命令。
镜像启动命令需要引用输入、输出参数中的变量,并以大括号扩起,以$符号进行引用。
镜像启动命令支持多行输入,每行最多256字符,最多支持300行。
镜像启动命令如下所示。
bash ${multifasta} -i ${indir} -o ${outfile}图30 选择镜像以及配置镜像启动命令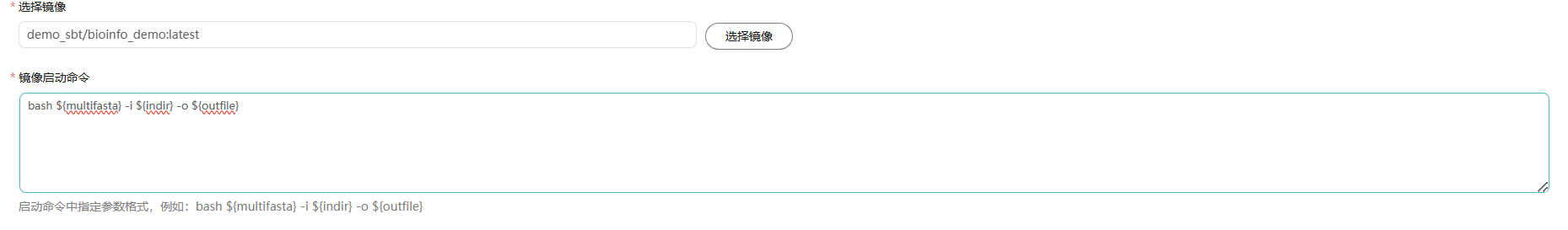
- 填写输入参数、输出参数。
参数填写时,输入参数及输出参数有字符串(String),文件(File),文件夹(Directory),枚举(Enum)四种类型。
图31 输入、输出参数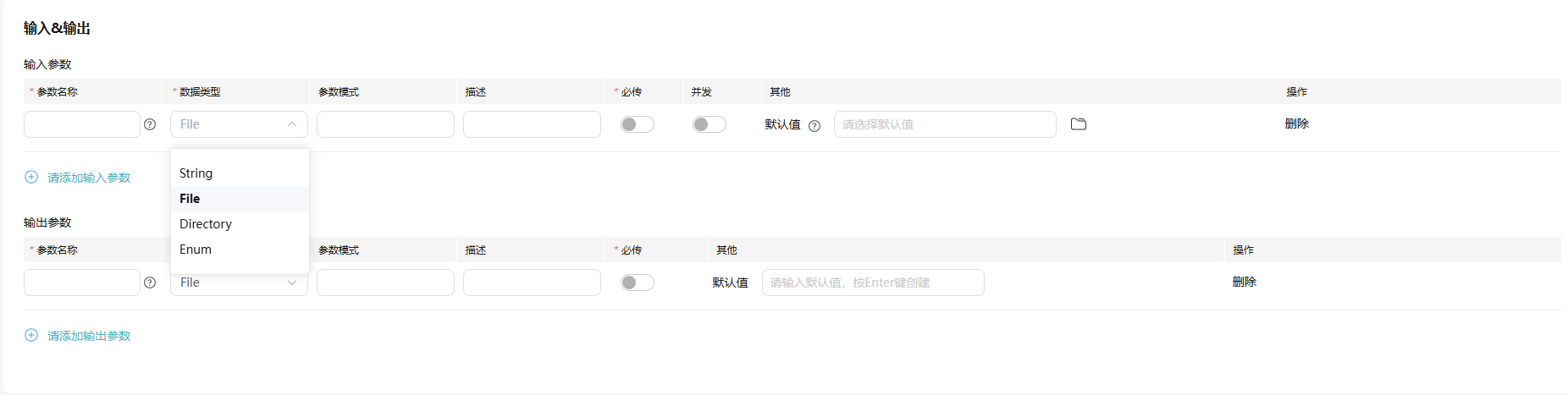

- 对于输入参数,打开“并发”开关,在启动作业时,每个参数可以设置多个参数值,自动生成多个作业并发执行。并发执行的作业数为设置的参数值个数的乘积。
例如,存在输入参数a和输入参数b,在启动作业时,分别给参数a设置了2个参数值,给参数b设置了2个参数值。那么,系统将自动生成4个作业并发执行。
- 对于输出参数,如果镜像启动命令中指定了输出参数,则在设置输出参数时,需要勾选“必传”,并填写“默认值”。
- 如果输出参数为String,默认值最大长度不超过256,仅支持字母、数字、中划线、下划线、小数点和斜线。
- 如果输出参数为Enum类型,需填写有效值,并在有效值中选择默认值。
- 对于输入参数,打开“并发”开关,在启动作业时,每个参数可以设置多个参数值,自动生成多个作业并发执行。并发执行的作业数为设置的参数值个数的乘积。
- 填写资源参数。
填写CPU、Memory大小(内存单位为GB),选择GPU类型。
- CPU架构依赖于制作镜像过程中选择的系统类型,以及制作镜像时所需的生物信息学软件支持在X86还是ARM上运行。例如,GATK是基于X86指令集开发的生信软件,使用CentOS的X86系统创建GATK镜像,则在创建应用时选择“X86”。
- CPU需求:请按实际需求填写,取值范围为“0.1-128”,单位C,支持一位小数,不填默认1C。
- Memory需求:请按实际需求填写,取值范围为“0.1-3072”,单位GB,支持一位小数,不填默认1GB。
- GPU类型:请按实际需求填写,取值范围为“无、GPU、Snt9”,如果选择Snt9,GPU需求需要是0、1、2、4、8。
- 计算节点标签:请选择标签名称,不支持多选。应用将会调度到有相应节点标签的计算节点。计算节点标签设置方法请参见《用户指南》的计算资源标签管理章节。
图32 资源参数
- 单击“立即创建”,创建应用。
步骤六:搭建流程、运行作业
- 单击“创建流程”,进入流程设计器页面。
图33 创建流程
- 将左侧应用列表中的应用,拖拽至画布中。
图34 应用拖拽
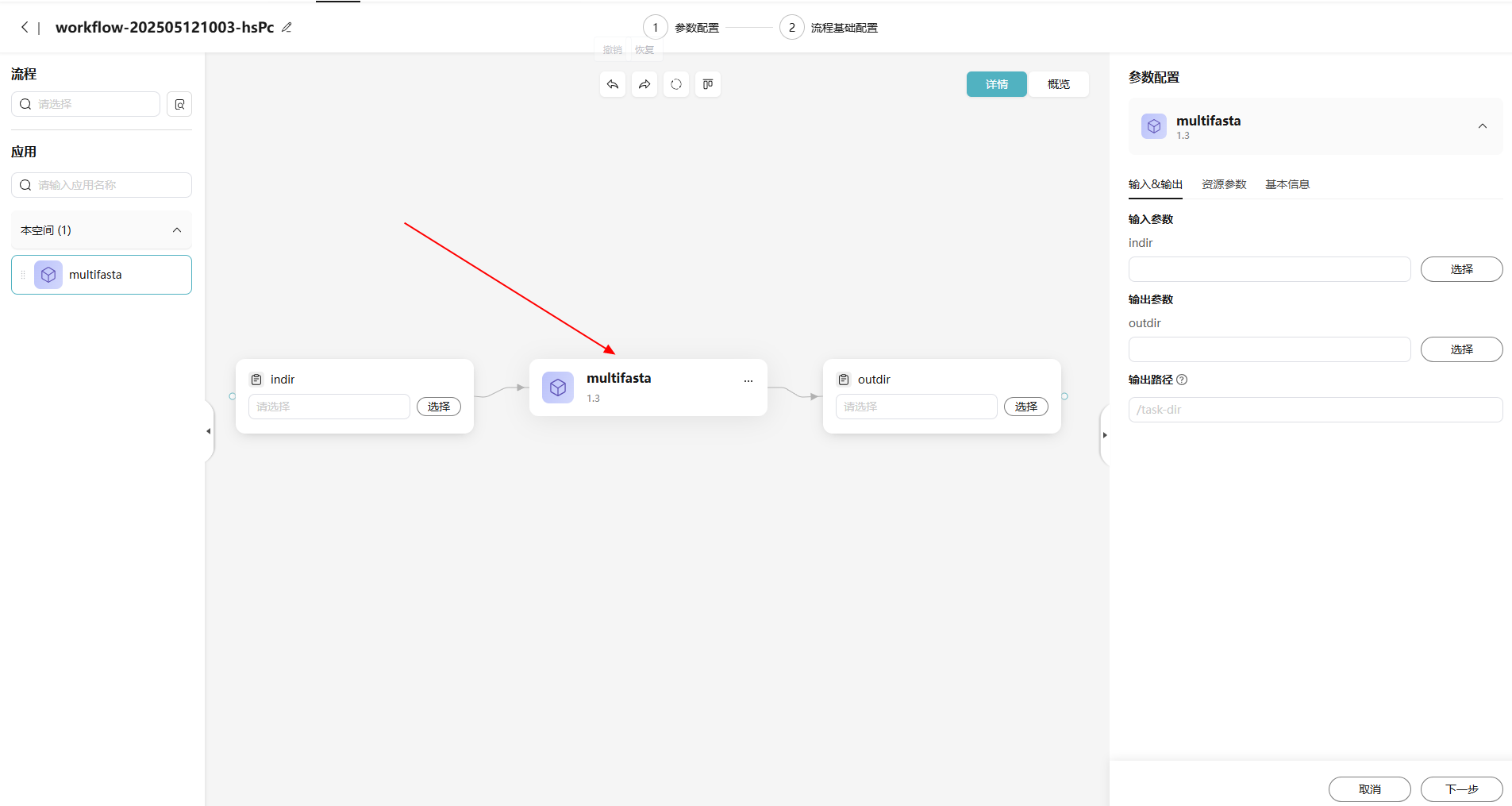
- 在画布右侧参数配置模块,单击
 按钮,进入参数设置页面,可查看修改参数。
按钮,进入参数设置页面,可查看修改参数。
- 输入参数:由用户在创建应用时定义。
- 输出路径:输出数据的存放路径,可修改。例如,gene-assets空间中的output文件夹,输出路径格式为/gene-assets/output。
- 资源参数:CPU需求、Memory需求、GPU类型,请按照使用需求进行设置;CPU架构、计算节点标签,GPU需求不可修改。GPU包含NAIDIA架构GPU和自研的Snt9+ARM。选择GPU资源时,需要您在购买平台时包含了GPU资源,并且需要应用支持GPU运行。选择Snt9+ARM时,需要应用支持ARM环境运行,并在创建应用时,镜像系统为ARM类系统。

运行分析作业时,流程中的每一个应用称之为一个任务(Task),在编排流程时,如果“输出路径”修改为空,实际作业输出结果依然默认存在该路径。最终输出结果路径按照如下规则生成:
- 若使用的应用本身有配置输出参数,直接使用该路径。
- 若使用的应用本身未配置输出参数,并且子任务(Task)的输出路径为空,最终输出路径为对应作业配置的输出路径。若作业也未配置,则系统会默认生成格式为job-流程名称-时间戳-随机后缀的输出路径,例如/job-workflowname-202501031644-1654。
- 若使用的应用本身未配置输出参数,子任务(Task)的输出路径有配置,例如/task-output。那么会在上一项的基础上拼接上子任务的输出路径,例如/job-workflowname-202501031644-1654/task-output。
图35 参数配置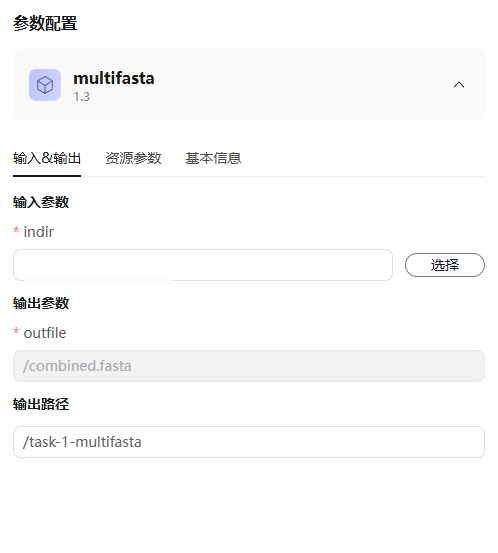
- 单击“下一步”,进入流程基础配置页面。
- 在流程基础配置页面填写流程基础信息。
包括“流程名称”、“版本”、“标签”、“短描述”、“长描述”和“超时时间”。
图36 流程基础配置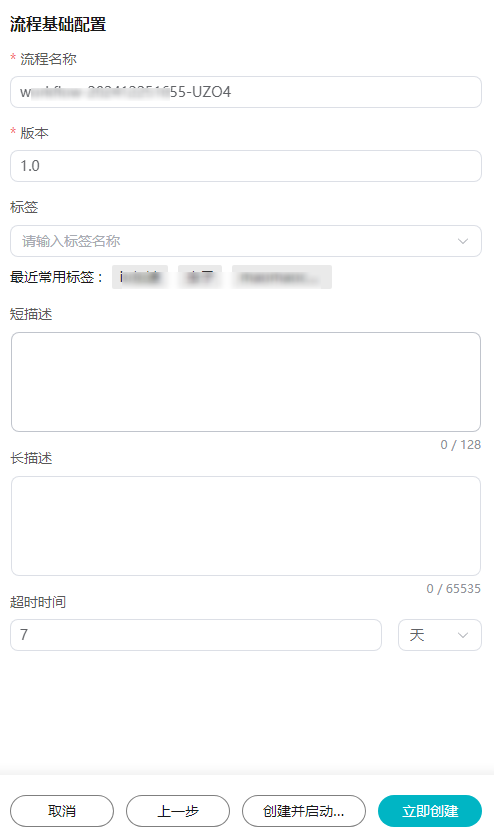

- “流程名称”和“版本”为必填项,其他参数可选填。
- “超时时间”指作业运行时间超过设置时间时即为超时,默认7天,基于流程创建分析作业时,该参数可重新定义。
- 单击“创建并启动作业”。