读取文件控制并发
在基因数据处理流程中,经常需要读取某个文件的内容来控制并发任务,或者获取另一个步骤的“输出结果”来控制并发任务。如,把样本文件按照固定大小进行拆分之后,需要得到所有的拆分文件名集合。或者上一步是分布式处理的,需要得到结果的总和。
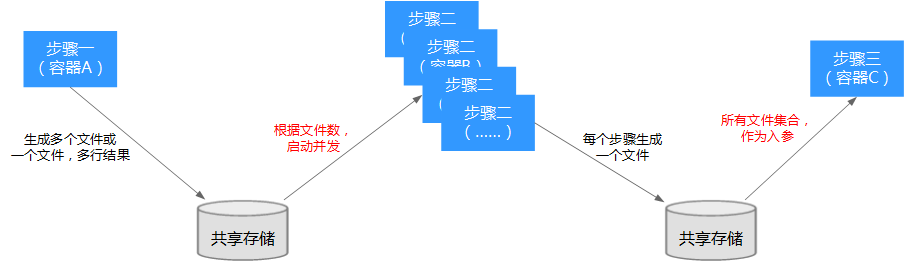
这种情况下,我们需要使用GCS语法中特定的功能,get_result函数来获得。
确定性的并发任务,GCS语法如下:
job-2:
commands_iter:
command: echo ${1} ${2}
vars_iter:
- [A, B, C] # <==== 注意这里,表示${1} 的并发数量(范围)
- [0, 1]
上述GCS语法中,[A, B, C] 表示并发数量为3数量。而动态的并发,就是数组[]中的值是上一步结果 。如下:
job-2:
commands_iter:
command: echo ${1} ${2}
vars_iter:
- get_result(job-1) # <==== 注意这里,数组结果是根据指定任务的stdout动态“计算出来的”
- [0, 1]
真正get到的result是指定步骤的标准输出,即“stdout”。
例如 job-1 的stdout为“1 2 3 4”,那么上一步的结果就是
vars_iter:
- ["1 2 3 4"]
注意,这里数组里面只有一个成员。也就是只有一个并发。
如果希望,每一个作为并发,可以加入“分割符”进行切分。具体语法见get_result函数。
vars_iter:
- get_result( job-1, " ")
会得到:
vars_iter:
- ["1", "2", "3", "4"]
这样就得到了4个并发,每个变量分别为"1", "2", "3", "4"。其中,分隔符是是任意的字符串。
在拥有get_result功能之后,如果需要实现根据上一步的结果,动态并发执行任务,则可以通过如下方式完成:

这样可以通过执行自己熟悉的shell命令,读取某个文件内容,或者列出目录中特定的文件,来得到想要的并发行为。
示例
job-list:
type: GCS.Job
tool: nginx:new-latest
commands: # <== (1) 列出目录中文件
- |
for i in `ls ${sfs}/${output-folder}`; do
echo ${i}
done
job-a:
type: GCS.Job
tool: nginx:new-latest
commands_iter:
command: echo ${output-prefix}job-c-${item} >> ${sfs}/${output-folder}/${1}; # <== (3) 迭代式并发,替换变量 ${1}
vars_iter:
- get_result(job-list, '\n') # <== (2) 把job-list输出,按一行一个,切分为并发数组
depends:
- target: job-list
type: whole







